はじめに : R版Slack API
RのSlack APIであるslackrパッケージの使い方について、いろいろとまとめてみた*1。
APIの諸設定、基本的なslackrの使い方に加えて、新着論文情報をRからチャネルに送信するプログラムも実装してみた。
まずは、Salck API設定の手順からはじめよう。
Salck API設定の手順
1. Slackのワークスペースに、任意のチャネルを作る。(必要なら)
2. Slack アプリを新規作成する
Rからは以下の関数実行で、Slack APIのサイトに飛べる。
browseURL("https://api.slack.com/apps/new")

ここで、任意のApp Nameを記入して、Development Slack Workspaceを選択する*2。
3. Incomming WebhooksをActiveにする
はじめに、Incomming Webhooks の設定に進む。
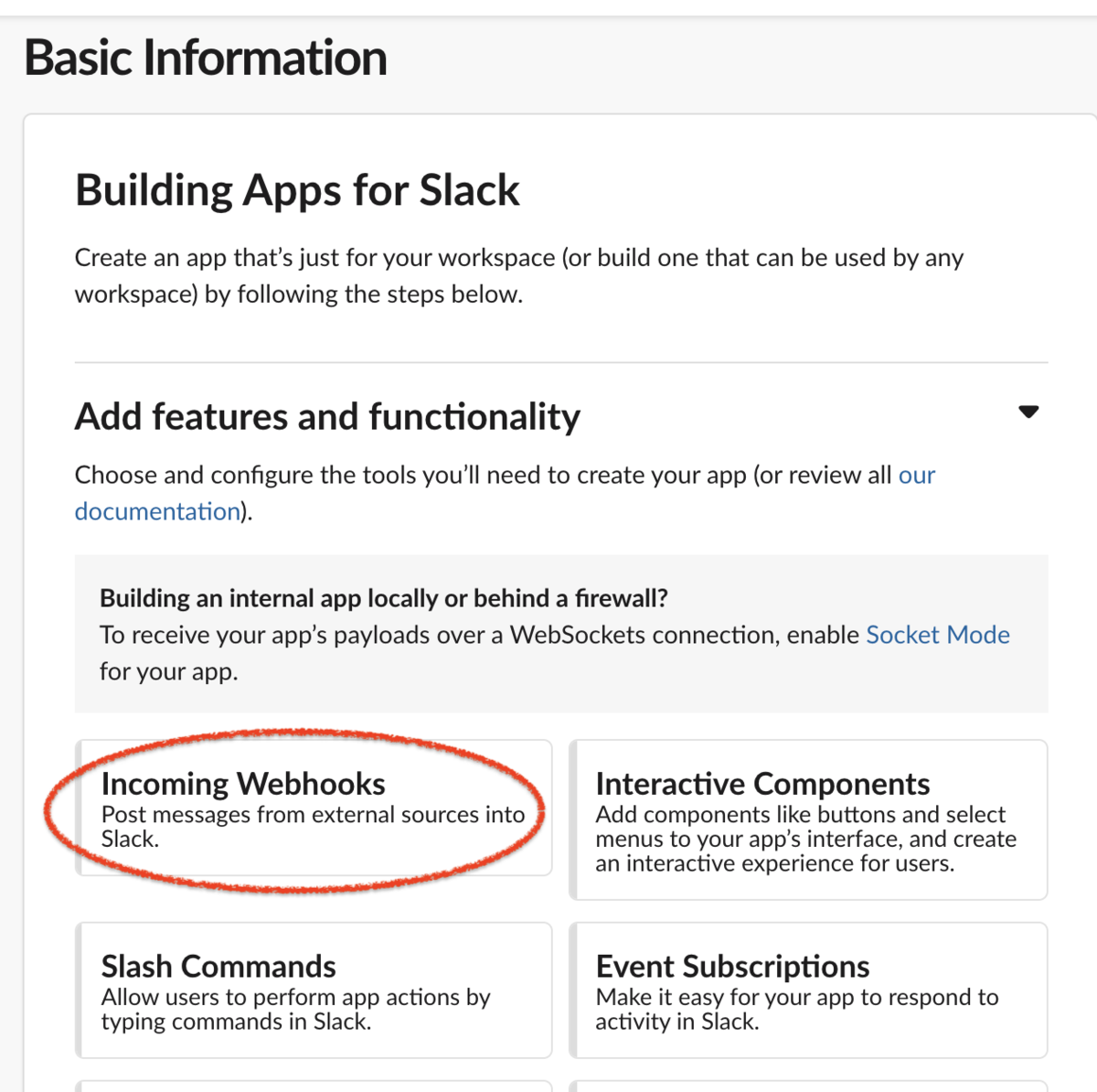
デフォルトでは、その設定はOFFになっている。
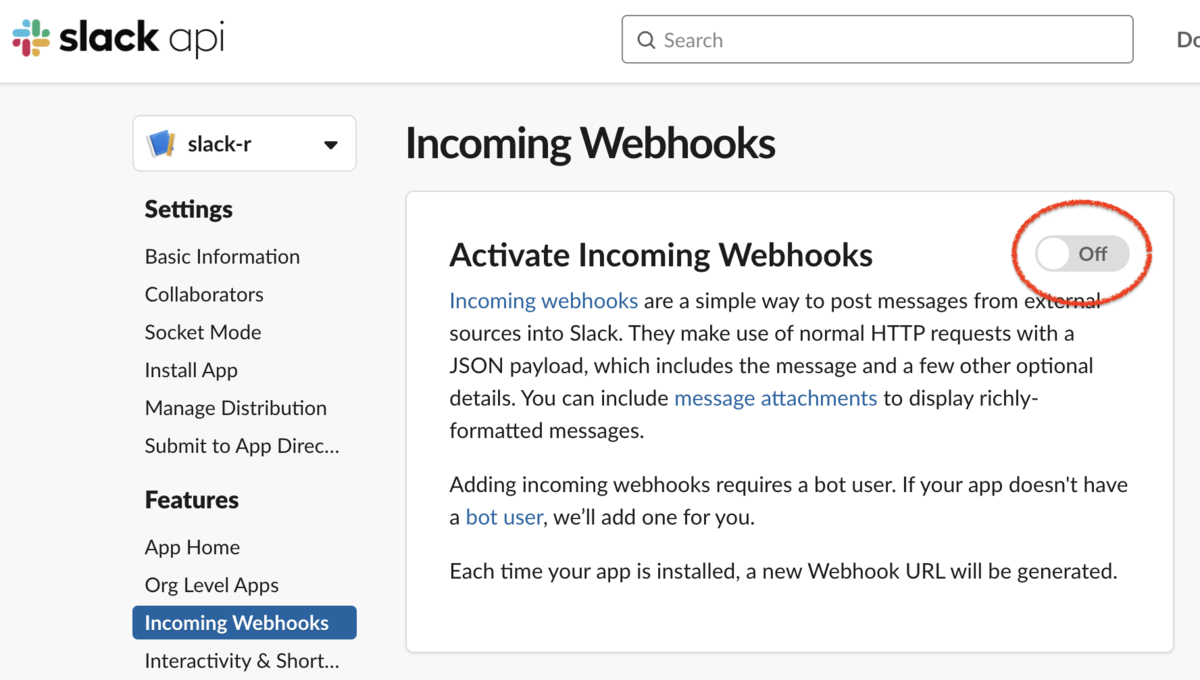
その設定を、ONにする。
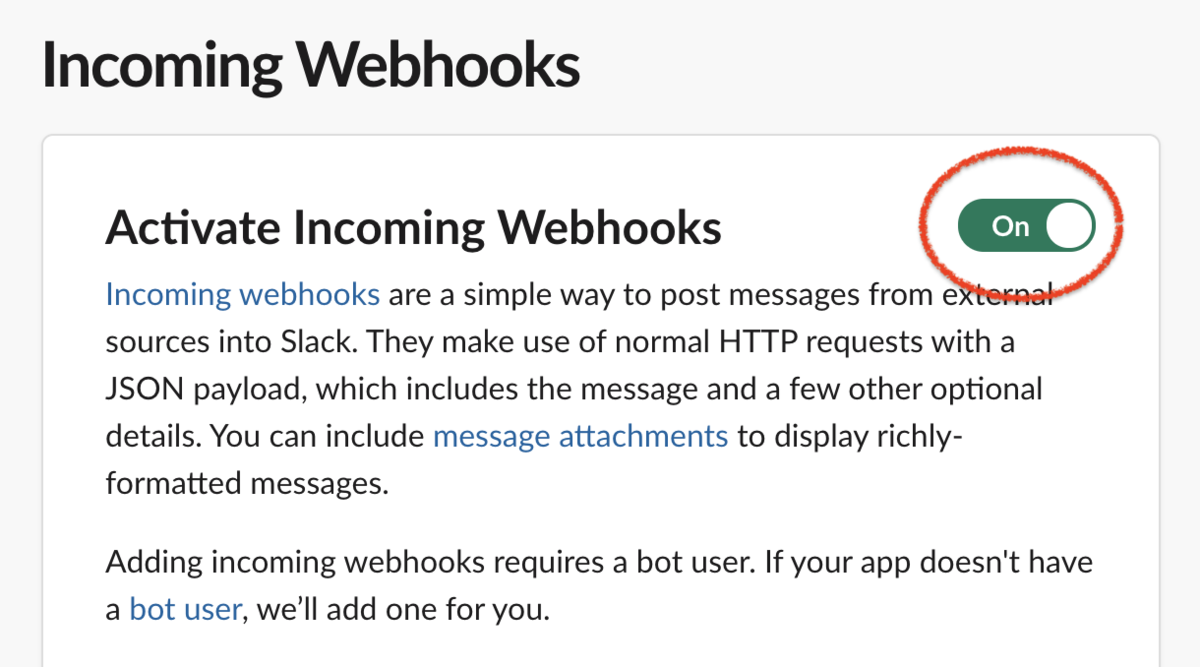
次に、Add New Webhook to Workspaceをクリックする

次の画面で、対象となるチャネルを選択して、許可する。
そうすると、Webhook URLが作成される。

ただ、この後のBot Token Scopesを設定して、reinstallすると、新たなWebhook URLが作成されるっぽいので、ひとまずはスルーしておく。
4. Bot User OAuth Tokenの取得、OAuth & Permissionsの設定
次に、サイドバーで、OAuth & Permissionsをクリックする。
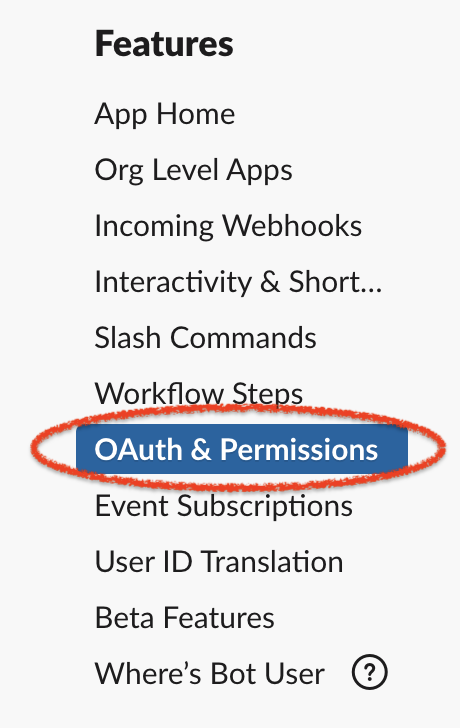
ここで、上部に表示される、Bot User OAuth Token は後ほど使用するので、コピーして、保存しておく。
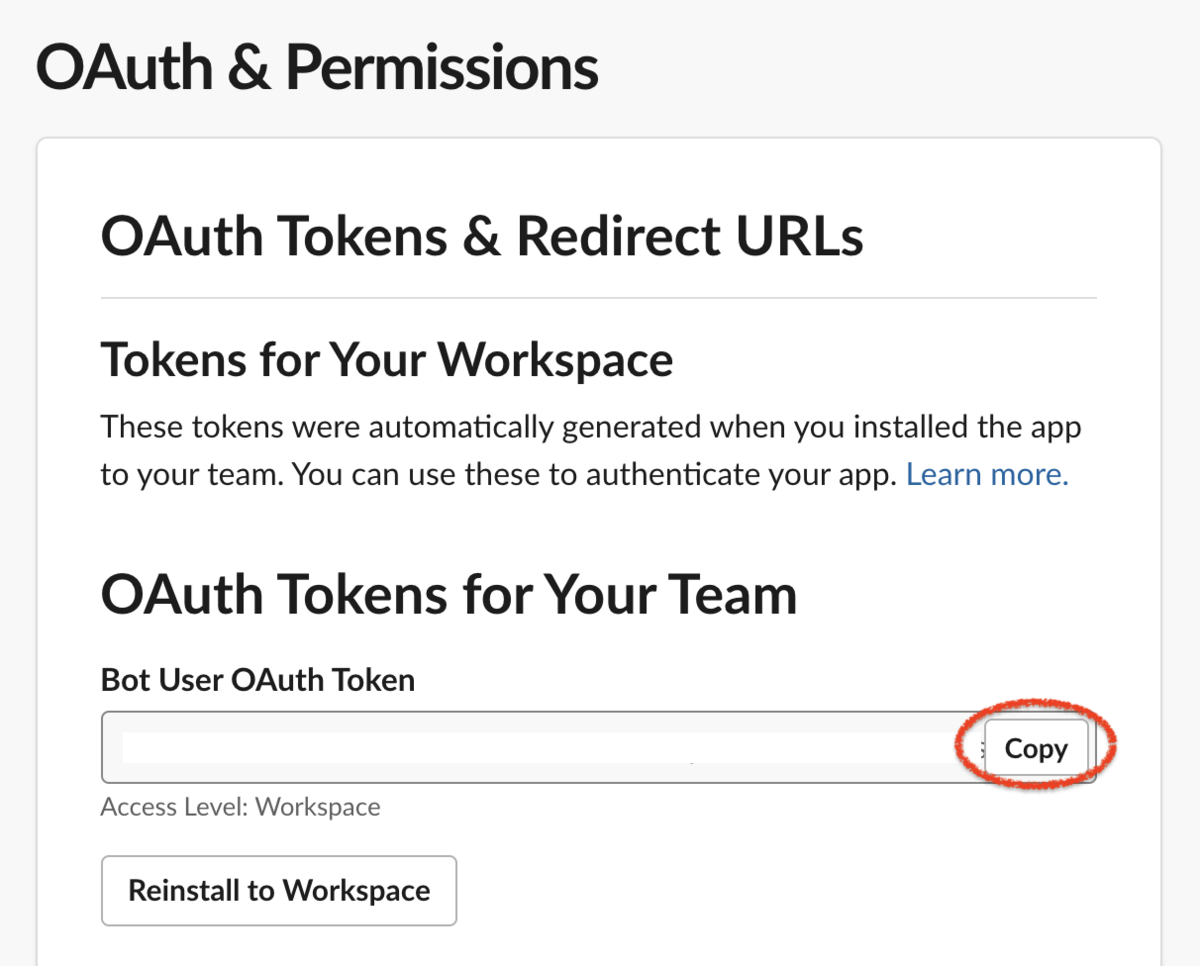
続いて、Bot Token Scopesを設定する。
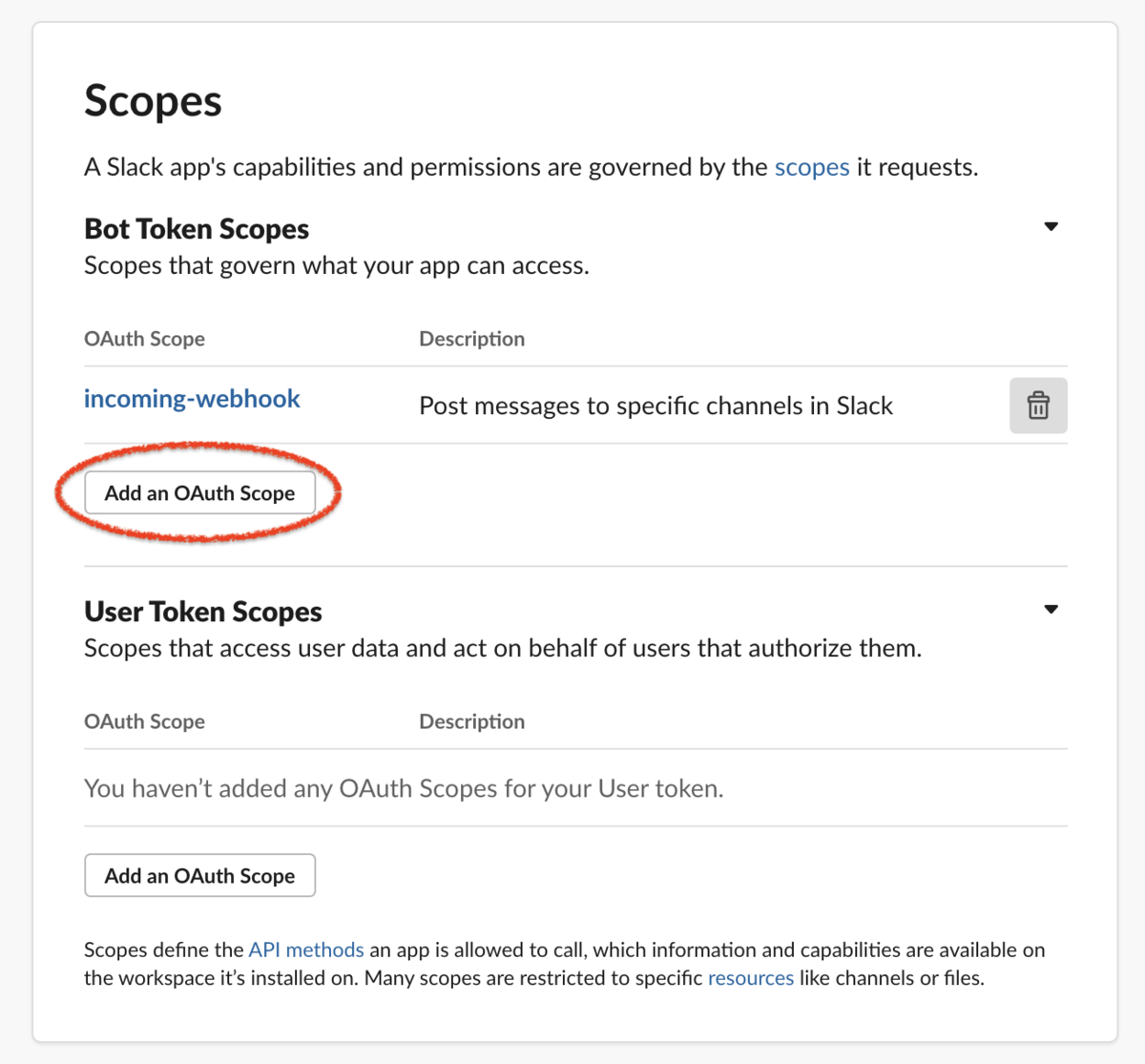
ここには、incoming-webhookのみが追加されているので、「Add an OAuth Scope」でScopeを追加する。
まずは、以下の項目あたりを追加して、様子をみるのが良い。
channels:readchat:writechat:write.customizechat:write.publicusers:readfiles:readfiles:writegroups:readgroups:writeim:readim:writechannels:history
上記が選択できたら、reinstallして、次の画面で、対象となるチャネルを選択して、許可する。
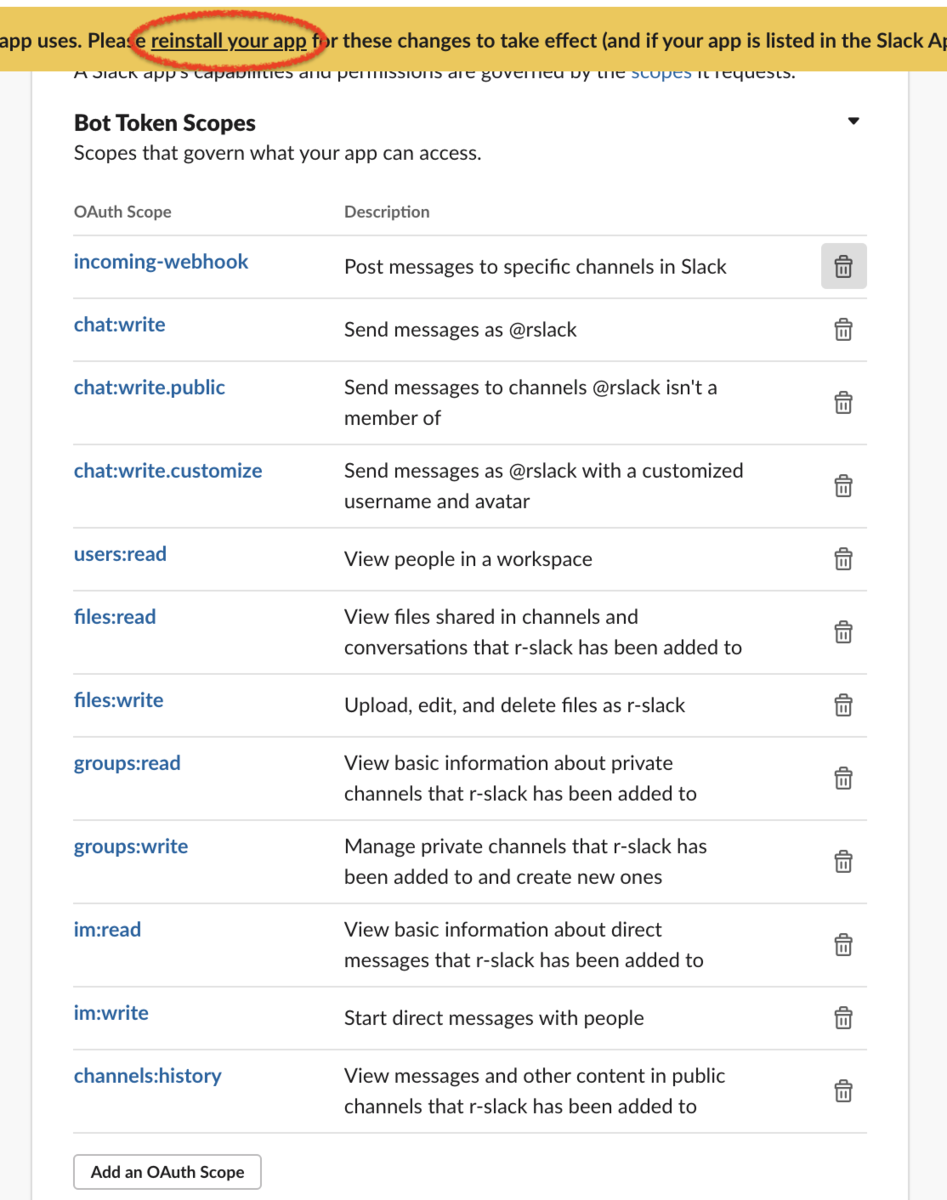
そうしたら、Incomming Webhooksの設定に戻って、新しく生成されたWebhook URLを取得しておく。

Sample curl requestのコードの最後部分が最新のWebhook URLなので、それをコピーしておく。
5. Slack アプリをチャネルに招待する【これが重要!】
どうも、以下の設定は必須みたい。
Known Issues
Depending on your scopes, slackr could quietly fail (i.e. not throw an error, but also not post anything to your channel). If this happens, try explicitly adding the app you’re trying to have slackr post as to the channel you want in your Slack workspace with /invite @your_app_name or make sure you have chat:write.public enabled.
日本語への機械翻訳: 既知の問題
スコープによっては、slackrが静かに失敗することがあります(エラーをスローせず、チャンネルに何もポストしないなど)。この問題が発生した場合は、Slackrに投稿させようとしているアプリをSlackのワークスペースに/invite @your_app_nameで明示的にチャンネルに追加するか、chat:write.publicを有効にしてください。
これは、chat:write.publicを有効にするだけでは解決しない。
やることは、対象チャネルで、「/invite @App Name」を入力して、作成したSlack アプリをチャネルに加える。
以下は、「@r-slack」を追加したときの例である。

送信すれば、「@r-slack」がチャネルに招待される。
6. RでのSlack APIの設定
ようやく、slackr側の設定を行う。
Rを起動して、まずは関連パッケージをインストール・ロードする。
#パッケージのインストール library(devtools) devtools::install_github("hrbrmstr/slackr", force = TRUE) library(slackr)
7. slackr config ファイルの設定
ここでは、Rからホームディレクトリに、~/.slackrを作成する。
上記で、コピー・保存しておいたOAuth TokenやWebhook URLを使用する。
.slackrの基本フォーマットは以下の通りである。
#基本的なフォーマット bot_user_oauth_token: Bot User OAuth Token channel: #チャネル名 username: 上記のApp Name incoming_webhook_url: https://hooks.slack.com/services/XXXXX/XXXXX/XXXXX
以下、R上から~/.slackrの作成を行う。tokenやチャネル名は、適時変更のこと。
また、~/.slackrを設定さえすれば、次回使用時からはslackr_setup()だけで完結する。
#Token & チャネル名 & Username の設定 OAuth_token <- "xoxb-XXXXXXX-XXXXXXX-XXXXXXX" WebURL <- "https://hooks.slack.com/services/XXXXX/XXXXX/XXXXX" Channel <- "チャネル名" #チャネル名は、"Channel ID"でも可 Username <- "slack-r" #Usernameは任意でOK a <- paste0(" bot_user_oauth_token: ", OAuth_token, " channel: ", Channel, " username: ", Username, " incoming_webhook_url: ", WebURL) #作成 system(paste0("echo \"", a, "\" > ~/.slackr")) #確認 system("cat ~/.slackr") #削除 (必要なら) #system("rm -rf ~/.slackr") #slackr_setupの実行 slackr_setup() #[1] "Successfully connected to Slack"
あと、オプション的な内容である。
#authentication & identity のチェック auth_test() #Slack usersの表示 slackr_users() #Slack channelsの表示 slackr_channels() #設定状況の確認 Sys.getenv("SLACK_CHANNEL") Sys.getenv("SLACK_BOT_USER_OAUTH_TOKEN")
8. 簡単な投稿実行
次に、実際に、Slackrパッケージを使って見ていこう。
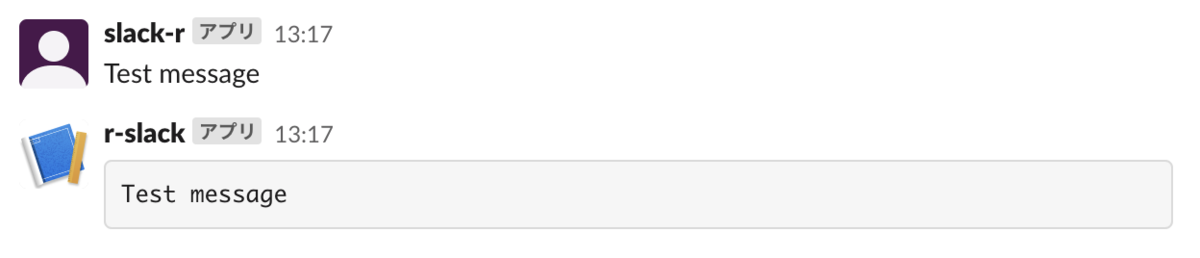
#メッセージ送信 slackr_msg("Test message") #メッセージをR表現の結果 「```形式」 として送信 slackr_bot('Test message')
上記の出力結果
#Rスクリプトの実行結果の送信 slackr(summary(UKgas)) #Plot結果を送信する plot(UKgas) slackr_dev(channels="#test") #OR ggslackr(plot(UKgas)) #画像のアップロード: 任意の画像で試してください。 slackr_upload("./01.png") #チャネルを指定して送信する場合 library(maps) map('county') slackr_dev(channels = "#test") #その他 #チャネルからメッセージを削除する #slackr_delete(count=1)
上記の出力結果(一部)

R/Slackrで、Google scholarで検索された新着論文情報を知らせる
応用例として、Google scholarで検索された論文情報を新着順に10件取得して、Slackのチャネルに投げるということをやってみる。
Rを用いたWebスクレイピングの詳細については、以下の記事を参考のこと。
また、googleScholarSearchNewest関数については、下記の補足資料を参照のこと。
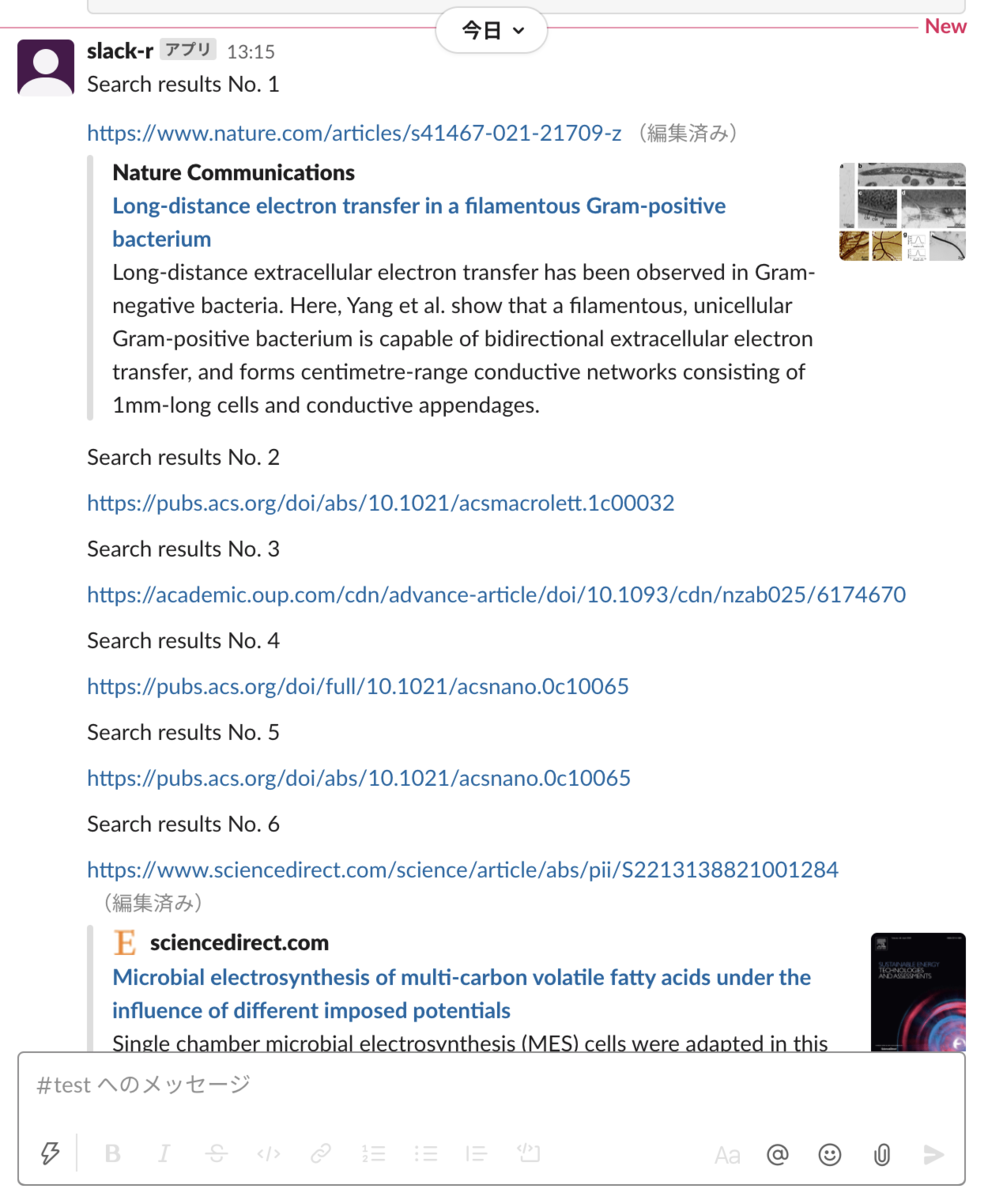
#パッケージとかの準備 if(!require("rvest")){install.packages("rvest")}; library(rvest) if(!require("xml2")){install.packages("xml2")}; library(xml2) if(!require("magrittr")){install.packages("magrittr")}; library(magrittr) source("https://gist.githubusercontent.com/kumeS/ef4d750a191b847174b3b93a4b8391c9/raw/9281e95919903fa66f2c43b71b9ae2340404d8a8/googleSearch_function.R") #検索語の準備 ex. electron microscopy Query <- c("electron microscopy") #Google Scholar 検索実行 d <- googleScholarSearchNewest(Query=Query) #結果ベクトルの取得 res <- d$`electron microscopy` res #[1] "https://www.nature.com/articles/s41467-021-21709-z" # [2] "https://pubs.acs.org/doi/abs/10.1021/acsmacrolett.1c00032" # [3] "https://academic.oup.com/cdn/advance-article/doi/10.1093/cdn/nzab025/6174670" # [4] "https://pubs.acs.org/doi/full/10.1021/acsnano.0c10065" # [5] "https://pubs.acs.org/doi/abs/10.1021/acsnano.0c10065" # [6] "https://www.sciencedirect.com/science/article/abs/pii/S2213138821001284" # [7] "https://link.springer.com/article/10.1140/epjp/s13360-021-01291-5" # [8] "https://pubs.acs.org/doi/abs/10.1021/acs.energyfuels.1c00167" # [9] "https://journals.sagepub.com/doi/abs/10.1177/0095244321996396" #[10] "https://www.sciencedirect.com/science/article/pii/S0269749121005315" #Slackへの出力 for(n in 1:length(res)){ slackr_msg(paste0("Search results No. ", n)) slackr_msg(res[n]) }
Slack上に、こんな感じで出力される
まとめ
ファイルのアップロードとか、スクリプトの情報共有とかは楽なのかもしれない。
今のところ、メッセージ送信とかレスポンスとかを、Rからすべてやるのはちょっとやり過ぎかもという所感である。