画像アレイデータを提供するexperimentHubパッケージ
BioImageDbsパッケージは、Rで可読なフォーマット(.Rds)で、 深層学習(DL)用の画像アレイデータ(4次元あるいは5次元)を 提供するexperimentHubパッケージである。
今回、BioImageDbsパッケージの使用方法を解説する。
BioImageDbsのインストール
まずは、Bioconductor関連のパッケージも含めて、インストールする。
#インストール install.packages("BiocManager") BiocManager::install("ExperimentHub") BiocManager::install("BioImageDbs") install.packages("magick") #ロード library(ExperimentHub) library(BioImageDbs)
BioImageDbsのデータセット表示
2021-05-18のスナップショットには、23 個のレコードが登録されています。
3D電顕解析で得られた神経系のデータセット、タイムラプスの細胞時系列データなどがあります。
#事前セットアップ eh <- ExperimentHub() #BioImageの情報取得 query(eh, "BioImageDbs") #ExperimentHub with 23 records # snapshotDate(): 2021-05-18 # $dataprovider: CELL TRACKING CHALLENGE (http://celltracki... # $species: Homo sapiens, Mus musculus, Drosophila melanoga... # $rdataclass: List, magick-image # additional mcols(): taxonomyid, genome, description, # coordinate_1_based, maintainer, rdatadateadded, # preparerclass, tags, rdatapath, sourceurl, sourcetype # retrieve records with, e.g., 'object[["EH6095"]]' # # title # EH6095 | EM_id0001_Brain_CA1_hippocampus_region_5dTensor... # EH6096 | EM_id0001_Brain_CA1_hippocampus_region_5dTensor... # EH6097 | EM_id0002_Drosophila_brain_region_5dTensor.rds # EH6098 | EM_id0002_Drosophila_brain_region_5dTensor_trai... # EH6099 | LM_id0001_DIC_C2DH_HeLa_4dTensor.rds # ... ... # EH6113 | LM_id0003_Fluo_N2DH_GOWT1_5dTensor.rds # EH6114 | EM_id0003_J558L_4dTensor.rds # EH6115 | EM_id0003_J558L_4dTensor_train_dataset.gif # EH6116 | EM_id0004_PrHudata_4dTensor.rds # EH6117 | EM_id0004_PrHudata_4dTensor_train_dataset.gif
画像アレイデータのダウンロード
(1) EM_id0001_Brain_CA1_hippocampus_region
画像セグメンテーション用の2値化ラベル付き5次元アレイ。 電子顕微鏡(FIB-SEM)画像の3Dセグメンテーションデータセット(1065x2048x1536ボリューム、約5x5x5nmの解像度)で、 マウス脳のCA1海馬領域にあるミトコンドリアのバイナリーラベル付き画像を含む。
#EM_id0001の情報取得 qr <- query(eh, c("BioImageDbs", "EM_id0001")) #タイトルの表示 mcols(qr)$title #[1] "EM_id0001_Brain_CA1_hippocampus_region_5dTensor.rds" #[2] "EM_id0001_Brain_CA1_hippocampus_region_5dTensor_train_dataset.gif" #詳細情報 qr[1] #ExperimentHub with 1 record # snapshotDate(): 2021-05-18 # names(): EH6095 # package(): BioImageDbs # $dataprovider: https://www.epfl.ch/labs/cvlab/data/data-em/ # $species: Mus musculus # $rdataclass: List # $rdatadateadded: 2021-05-18 # $title: EM_id0001_Brain_CA1_hippocampus_region_5dTensor.rds # $description: 5D arrays with the binary label for the ima... # $taxonomyid: 10090 # $genome: NA # $sourcetype: PNG # $sourceurl: https://github.com/kumeS/BioImageDbs # $sourcesize: NA # $tags: c("3D images", "bioimage", "CellCulture", # "electron microscopy", "microscope", "scanning # electron microscopy", "segmentation", "Tissue") # retrieve record with 'object[["EH6095"]]' #アレイデータのダウンロード ImageData <- qr[[1]] #アレイデータの詳細表示 str(ImageData$Train) #List of 2 # $ Train_Original : num [1, 1:1024, 1:768, 1:165, 1] 0.4 0.518 0.471 0.537 0.51 ... # $ Train_GroundTruth: num [1, 1:1024, 1:768, 1:165, 1] 0 0 0 0 0 0 0 0 0 0 ... str(ImageData$Test) #List of 2 # $ Test_Original : num [1, 1:1024, 1:768, 1:165, 1] 0.569 0.514 0.475 0.482 0.533 ... # $ Test_GroundTruth: num [1, 1:1024, 1:768, 1:165, 1] 0 0 0 0 0 0 0 0 0 0 ... #動画データ(.gif)の表示 magick::image_read(qr[[2]])
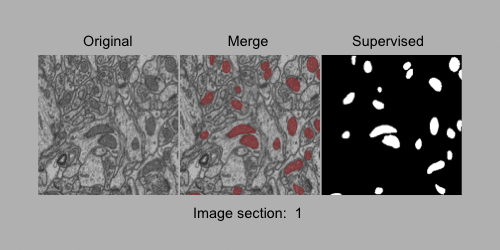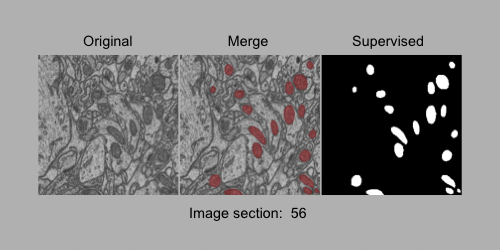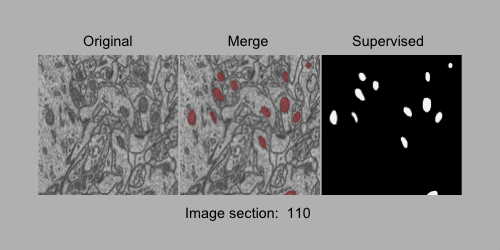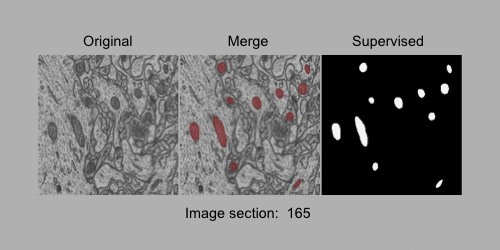
ここで、qr[]でメタデータにアクセスできる。
また、qr[[]]で実データにアクセスできる。
(2) LM_id0001_DIC_C2DH_HeLa
2D画像セグメンテーション用のHeLa細胞の4次元アレイデータである。 オリジナルデータとマルチラベルが付与された教師画像データのセットである。 データセットの画像は、対物レンズにPlan-Apochromat 63x/1.4(oil)を使用したZeiss LSM 510 Metaで撮影された。
#LM_id0001の情報取得 qr <- query(eh, c("BioImageDbs", "LM_id0001")) #タイトルの表示 mcols(qr)$title #[1] "LM_id0001_DIC_C2DH_HeLa_4dTensor.rds" #[2] "LM_id0001_DIC_C2DH_HeLa_4dTensor_train_dataset.gif" #[3] "LM_id0001_DIC_C2DH_HeLa_4dTensor_Binary.rds" #[4] "LM_id0001_DIC_C2DH_HeLa_4dTensor_Binary_train_dataset.gif" #[5] "LM_id0001_DIC_C2DH_HeLa_5dTensor.rds" #詳細情報 qr[1] #ExperimentHub with 1 record # snapshotDate(): 2021-05-18 # names(): EH6099 # package(): BioImageDbs # $dataprovider: CELL TRACKING CHALLENGE (http://celltrackingchallenge.net/2d-datasets/) # $species: Homo sapiens # $rdataclass: List # $rdatadateadded: 2021-05-18 # $title: LM_id0001_DIC_C2DH_HeLa_4dTensor.rds # $description: 4D arrays with the multi-labels for the image segmentation. Human HeLa ... # $taxonomyid: 9606 # $genome: NA # $sourcetype: PNG # $sourceurl: https://github.com/kumeS/BioImageDbs # $sourcesize: NA # $tags: c("bioimage", "cell tracking", "CellCulture", "microscope", # "segmentation", "Tissue") # retrieve record with 'object[["EH6099"]]' #アレイデータのダウンロード ImageData <- qr[[1]] #アレイデータの詳細表示 str(ImageData$Train) #List of 2 # $ Train_Original : num [1:84, 1:512, 1:512, 1] 0.518 0.455 0.455 0.447 0.439 ... # $ Train_GroundTruth: num [1:84, 1:512, 1:512, 1] 0 0 0 0 0 0 0 0 0 0 ... str(ImageData$Test) #List of 2 # $ Test_Original : num [1:84, 1:512, 1:512, 1] 0.604 0.467 0.459 0.435 0.408 ... # $ Test_GroundTruth: num [1:84, 1:512, 1:512, 1] 0 0.671 0.671 0.851 0.851 ... #詳細情報 qr[2] #ExperimentHub with 1 record # snapshotDate(): 2021-05-18 # names(): EH6100 # package(): BioImageDbs # $dataprovider: CELL TRACKING CHALLENGE (http://celltrackingchallenge.net/2d-datasets/) # $species: Homo sapiens # $rdataclass: magick-image # $rdatadateadded: 2021-05-18 # $title: LM_id0001_DIC_C2DH_HeLa_4dTensor_train_dataset.gif # $description: A animation file (.gif) of the train dataset of LM_id0001_DIC_C2DH_HeLa... # $taxonomyid: 9606 # $genome: NA # $sourcetype: PNG # $sourceurl: https://github.com/kumeS/BioImageDbs # $sourcesize: NA # $tags: c("animation", "bioimage", "cell tracking", "CellCulture", # "microscope", "segmentation", "Tissue") # retrieve record with 'object[["EH6100"]]' #動画データ(.gif)の表示 magick::image_read(qr[[2]])





