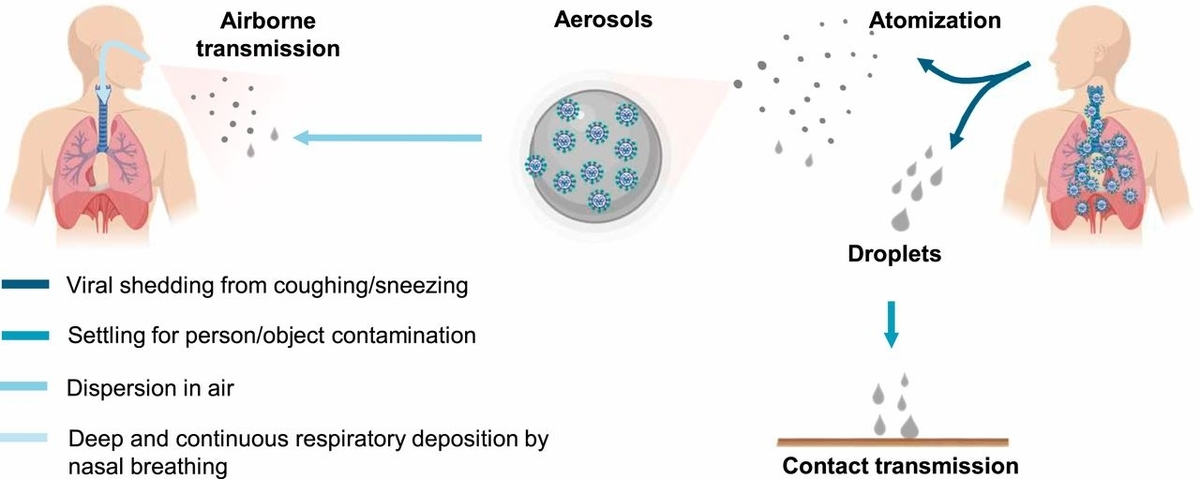
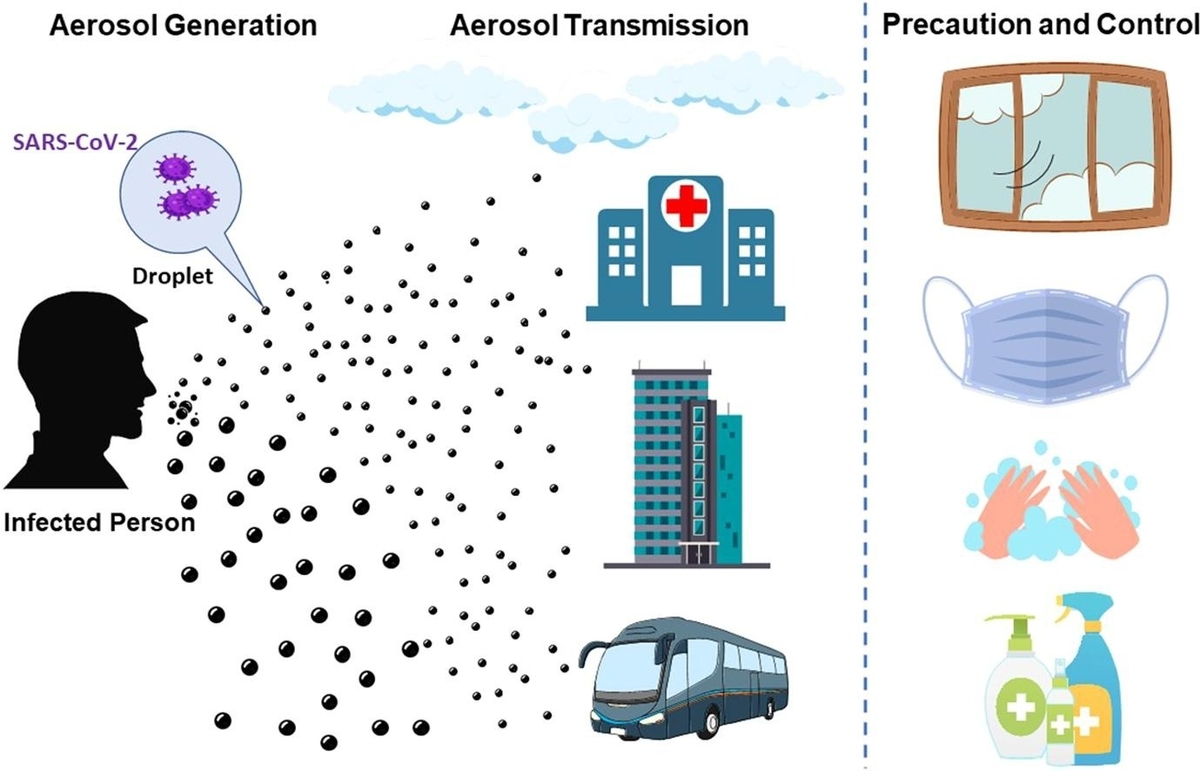
はじめに
この記事は、2021年12月30日現在のWikipedia 英語記事「COVID-19」を日本語訳したものである。
この情報は英語ページにはありますが、まだ日本語ページは存在していません。結構しっかりした内容だったので、このwikipedia記事の日本語訳を作成することにしました。
コロナ関連の情報はすぐに古くなりますので、その都度、新しい情報を確認してください。
Pathophysiology / 病態生理
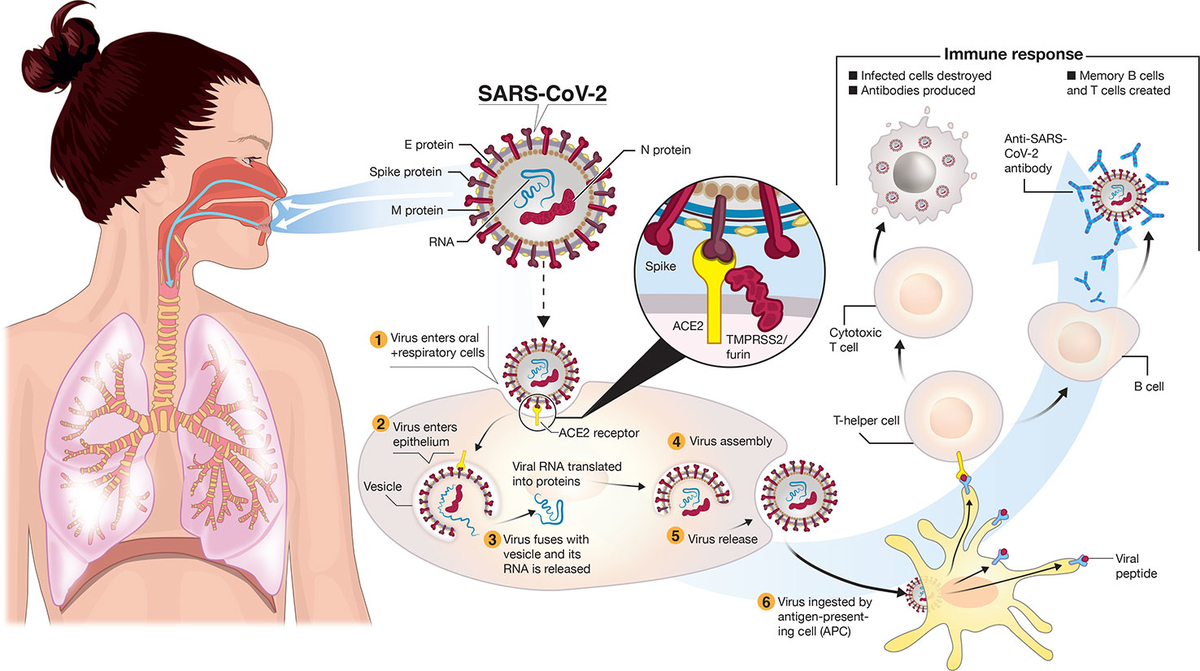
SARS-CoV-2ウイルス(COVID-19)は、生体内の様々な細胞やシステムに感染する。 COVID-19は、上気道(副鼻腔、鼻、喉)および下気道(気管および肺)に感染することがよく知られている*1。 肺は、COVID-19の影響を最も受ける臓器である。 なぜなら、ウイルスは、肺のII型肺胞細胞の表面に最も豊富に存在する、アンジオテンシン変換酵素2(ACE2)の受容体を介して、宿主細胞にアクセスする*2。 ウイルスは、「スパイク」と呼ばれる、特殊な表面糖タンパク質を用いて、ACE2受容体とコネクトして、宿主細胞に侵入する*3。

過去の記事
*1:Harrison AG, Lin T, Wang P (December 2020). "Mechanisms of SARS-CoV-2 Transmission and Pathogenesis". Trends in Immunology. 41 (12): 1100–1115. doi:10.1016/j.it.2020.10.004. PMC 7556779. PMID 33132005.
*2:Verdecchia P, Cavallini C, Spanevello A, Angeli F (June 2020). "The pivotal link between ACE2 deficiency and SARS-CoV-2 infection". European Journal of Internal Medicine. 76: 14–20. doi:10.1016/j.ejim.2020.04.037. PMC 7167588. PMID 32336612.
*3:Letko M, Marzi A, Munster V (April 2020). "Functional assessment of cell entry and receptor usage for SARS-CoV-2 and other lineage B betacoronaviruses". Nature Microbiology. 5 (4): 562–569. doi:10.1038/s41564-020-0688-y. PMC 7095430. PMID 32094589.
*4:感染症などによって、サイトカイン(IL-1,IL-6,TNF-αなど)が異常に上昇して、その作用が全身に及ぶことで、好中球の活性化、血液凝固機構活性化、血管拡張などを全身に起こり、多臓器不全にまで進行する。この状態を、サイトカインストーム(cytokine storm)という。