
はじめに
「R/Keras/TensorFlowでやるディープラーニングのすゝめ」の連載2回目です(2022年1月19日アップデート版)。
【1】では、ベクトルデータに対する Autoencoderを取り上げましたが、
今回は、 2D Convolutional Neural Network (CNN: 畳み込みニューラルネットワーク) を使ったAutoencoderの実装について概説します。
skume.net
下記で登場する、CNN、Maxプーリング、Upサンプリング、活性化関数
などについては、すでに多くの分かりやすい説明記事がありますので、そちらを参照のこと。
qiita.com
deepage.net
qiita.com
R/Kerasのセットアップ
RStudioを起動して、Kerasパッケージをロードします。そして、Pythonを選択します。
library(keras)
reticulate::use_python("/usr/local/bin/python", required =T)
R/Kerasのセットアップの詳細については、前回の記事とかを参照のこと。
skume.hatenablog.com
www.slideshare.net
MNISTデータの準備
今回も、手書きアラビア文字であるMNISTデータセットを使用します。
MNISTデータセットをロードして、リスト型からアレイ型に変換します。
同データセットのトレーニングデータは6万枚の画像りますがが、
それを全て使用すると、残念ながら、CPU計算ではなかなか収束しない・・・
そこで、トレーニングデータの内で、1000枚の画像をランダムで
ピックアップして、新たにデータセットを作ります。
また、2D CNNレイヤーを使用する場合、
inputデータは、4次元アレイ型(= 4次元テンソル)に変換しておくのがポイントです。
Data <- dataset_mnist()
str(Data)
xtrain <- Data$train$x
ytrain <- Data$train$y
xtrain <- xtrain/255
Sam <- sample(1:dim(xtrain)[1], 1000, replace = F)
xtrain <- xtrain[Sam,,]
ytrain <- ytrain[Sam]
x_train <- array_reshape(xtrain, dim=c(dim(xtrain)[1], 28, 28, 1))
dim(x_train)
このとき、4次元アレイ(= 4次元テンソル)は、
1次元目を画像番号、2と3次元目を2D画像データ、
4次元目をチャネル数( グレイスケールの場合、1 、RGBの場合、3 )で与えます。
Autoencoder with 2D CNN のモデル構築
2D CNNを使用する場合、実際の入力データは、4次元アレイの1次元目を除いた、2〜4次元目の3次元アレイとなります。
このアレイは、1と2次元目を2D画像データ、3次元目をチャネル数で与えます。
そのため、layer_inputのshapeは、c(28, 28, 1) と3要素ベクトル(ピクセル数とチャネル数の組み合わせ)で記述します。
ちょっとした関数の説明
layer_conv_2d関数で、2次元畳み込み層(例えば、画像上の空間畳み込み)を与える。
layer_conv_2dのfiltersは、出力空間の次元(すなわち、畳み込みの出力フィルタの数)を整数値で与える。
layer_conv_2dのkernel_size(sizeで与える)は、2次元畳み込みウィンドウの幅と高さを、整数値で与える。
layer_conv_2dのpaddingでは、"valid" か "same" を指定する。"same"の場合、同じアレイを返す。
layer_max_pooling_2d関数は、空間データの最大プーリング処理を行う層を与える。
layer_max_pooling_2dのpool_sizeは、ダウンスケールする係数 (垂直, 水平)を整数値で与える。例えば、c(2, 2) を指定すると、垂直・水平の空間次元で入力が半分になる。
layer_upsampling_2d関数は、2D入力用のアップサンプリング層を与える。
layer_upsampling_2dのsizeでは、行と列のアップサンプリング係数を整数値で与える。
これらのKeras内の関数を使って、autoencoderモデルを構築してみる。
input <- layer_input(shape = c(28, 28, 1))
kernel_size <- c(3,3)
filters <- 32
output = input %>%
layer_conv_2d(filters=filters, kernel_size=kernel_size, activation="relu", padding="same") %>%
layer_max_pooling_2d(pool_size=c(2,2), padding="same") %>%
layer_conv_2d(filters=filters, kernel_size=kernel_size, activation="relu", padding="same") %>%
layer_max_pooling_2d(pool_size=c(4,4), padding="same") %>%
layer_conv_2d(filters=filters, kernel_size=kernel_size, activation="relu", padding="same") %>%
layer_upsampling_2d(size=c(4,4)) %>%
layer_conv_2d(filters=filters, kernel_size=kernel_size, activation="relu", padding="valid") %>%
layer_upsampling_2d(size=c(2,2)) %>%
layer_conv_2d(filters=1, kernel_size=kernel_size, activation="sigmoid", padding="same")
Autoencoder2DCNN <- keras_model(input, output)
summary(Autoencoder2DCNN)
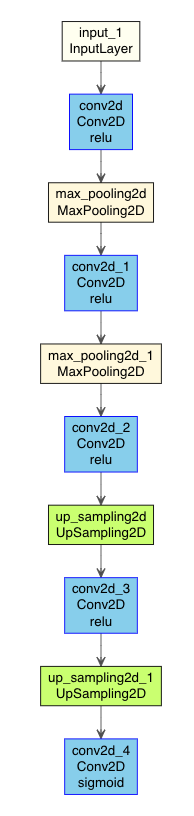
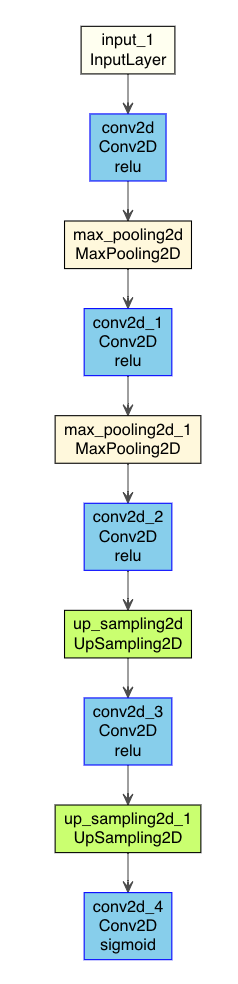
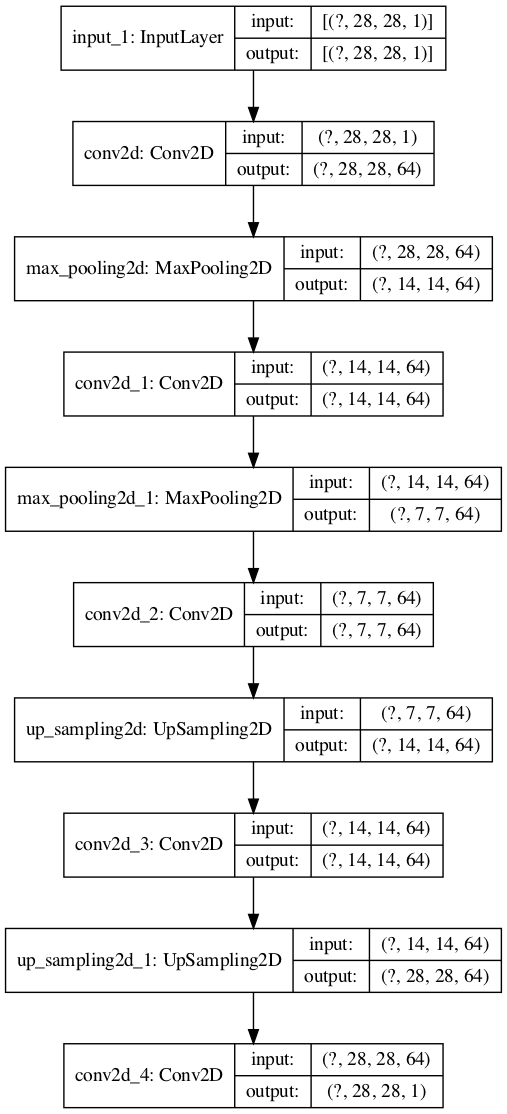
このとき、input_1 (InputLayer) [(None, 28, 28, 1)]と
conv2d_4 (Conv2D) (None, 28, 28, 1)のサイズを一致させる必要があります。
このモデルでは、28x28 => 14x14 => 4x4 => 16x16 => 14x14 => 28x28 とピクセルサイズがダウンスケール・アップスケールしています。
このとき、4x4以下までピクセル数を下げると、精度が悪くなる。また、16x16 => 14x14は、layer_conv_2dのpaddingを validとすることでピクセル数を微調整しています。
あと、MNISTデータは、28x28ピクセルであるが、モデル構築の観点からは、使用する2D画像のピクセル数は、2N x 2N ピクセル(例えば、25 = 32、27 = 128)が望ましいです。
DLモデルの出力
source("https://gist.githubusercontent.com/kumeS/41fed511efb45bd55d468d4968b0f157/raw/0f64b83700ac578d0c39abd420da5373d4317083/DL_plot_modi_v1.1.R")
Autoencoder2DCNN %>% plot_model_modi(width=1, height=1.25)
(plot_model_modiの出力結果)
TensorFlowでもやってみると
tf <- reticulate::import(module = "tensorflow")
py_plot_model <- tf$keras$utils$plot_model
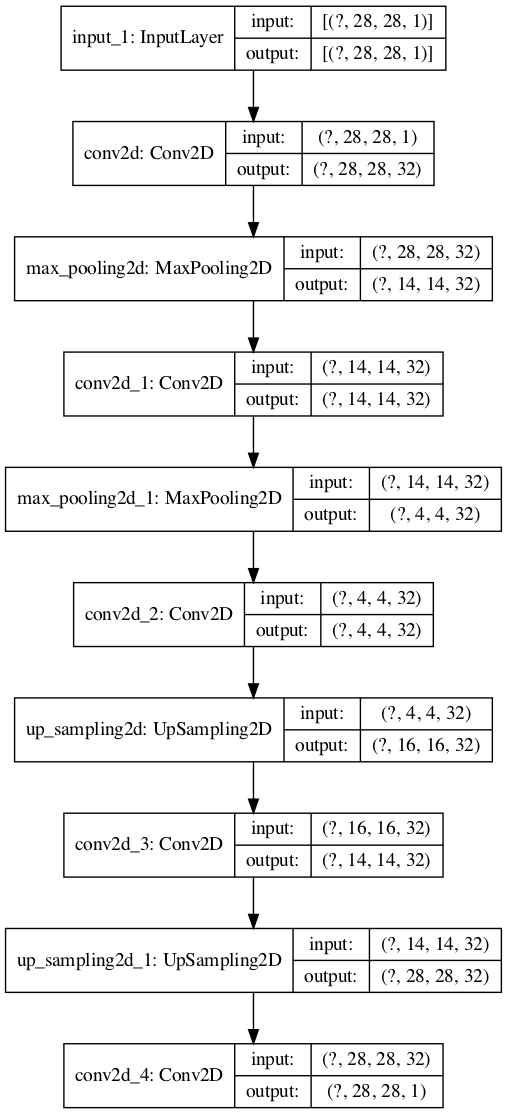
py_plot_model(Autoencoder2DCNN, to_file='Autoencoder2DCNN_tf.png',
show_shapes=T, show_layer_names=T,
expand_nested=T, dpi=100)
(py_plot_modelの出力結果)
モデルが確認できれば、compile & fit を実行する。
Autoencoder2DCNN %>%
compile(optimizer="rmsprop", loss="mean_squared_error")
Autoencoder2DCNN %>%
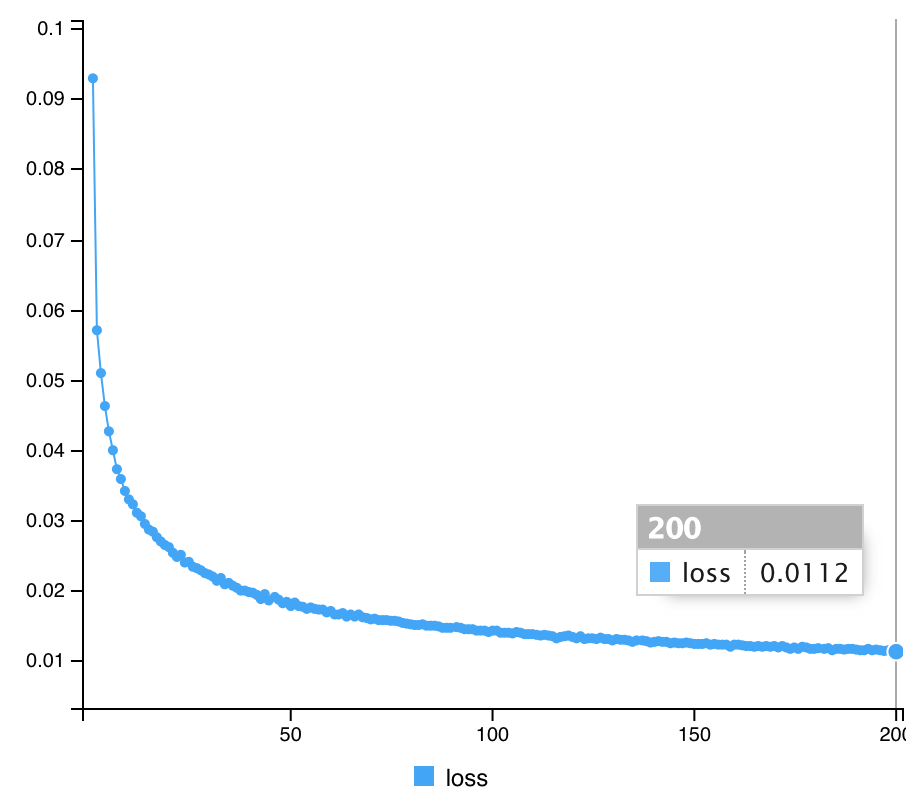
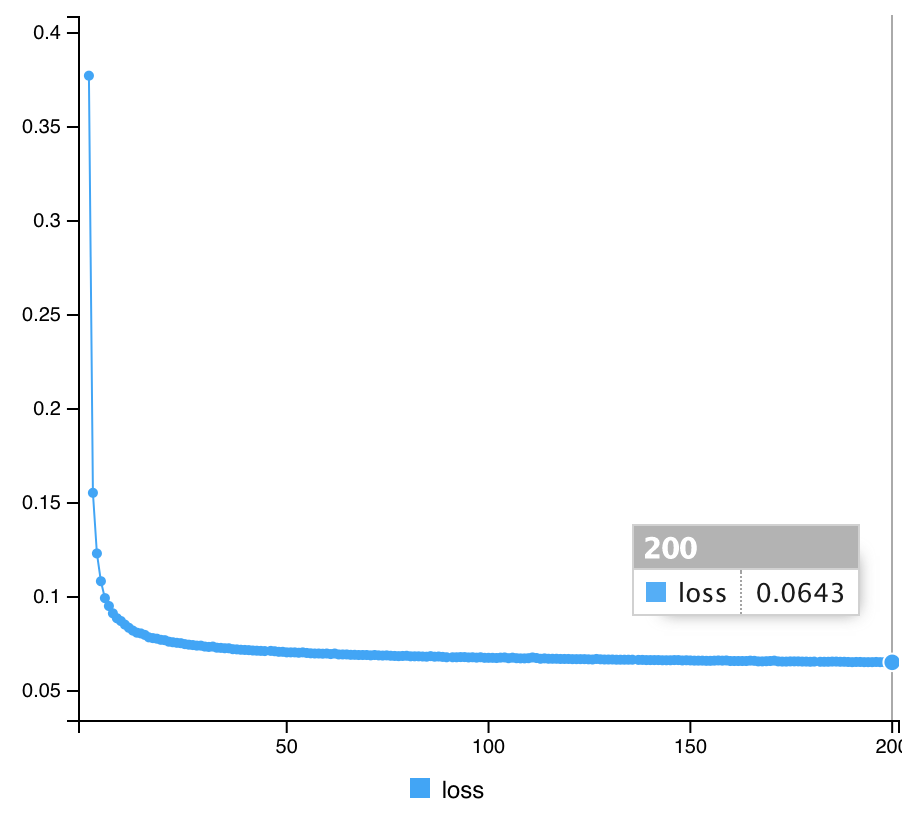
fit(x_train, x_train, epochs=200, batch_size=32,
shuffle = T, verbose=1)
(トレーニングの結果)
結果の評価
次に、このモデルによるTrainデータセットの変換結果を実際の画像と比較してみます。
まずは、アレイ型を4次元から3次元に変換します。
library(EBImage)
pred_imgs <- Autoencoder2DCNN %>% predict(x_train)
pred_imgsR <- array_reshape(pred_imgs, dim=c(dim(pred_imgs)[1], 28, 28))
dim(pred_imgsR)
par(mfrow=c(3,2))
for (i in 1:6) {
m <- sample(1:dim(xtrain)[1], 1, replace = F)
display(combine(t(xtrain[m,,]), t(pred_imgsR[m,,])),
method="raster", nx=2, all=TRUE, spacing = 0.01, margin = 2)
}
左側が入力画像(オリジナル画像)、右側が予測出力画像の結果を示します。

さらに、Autoencoderの圧縮特徴量層を取り出して、t分布型確率的近傍埋め込み法 (t-SNE; T-distributed Stochastic Neighbor Embedding) で次元圧縮を行い、2D 表示をしてみます。
summary(Autoencoder2DCNN)
intermediate_layer <- keras_model(inputs = Autoencoder2DCNN$input,
outputs = get_layer(Autoencoder2DCNN, "conv2d_2")$output)
summary(intermediate_layer)
intermediate_output <- predict(intermediate_layer, x_train)
str(intermediate_output)
intermediate_output_re <- array_reshape(intermediate_output, dim=c(dim(intermediate_output)[1], dim(intermediate_output)[2]*dim(intermediate_output)[3]*dim(intermediate_output)[4]))
xy <- data.frame(ytrain, intermediate_output_re)
library(Rtsne)
xy.tSNE <- Rtsne(as.matrix(xy[,-1]), check_duplicates = FALSE, verbose=TRUE,
dims = 2, perplexity = 25, theta = 0.5, max_iter = 5000)
plot(xy.tSNE$Y, cex=0.5, pch=19, col=rainbow(10)[xy[,1]+1],
xlab="t-SNE PC1", ylab="t-SNE PC2")
par(mfrow=c(1,1), mai=c(0.75,0.75,0.2,0.2), mgp = c(2,1,0))
xy1 <- data.frame(ytrain, xy.tSNE$Y)
plot(xy1[,2:3], cex=0.5, pch=19, col="white",
xlab="t-SNE PC1", ylab="t-SNE PC2")
a <- range(xy1[,2][is.finite(xy1[,2])])
b <- range(xy1[,3][is.finite(xy1[,3])])
a1 <- diff(a)*0.0125
b1 <- diff(b)*0.0125
for(n in 1:nrow(xy1)){
v <- col2rgb(rainbow(10)[xy1[n,1] + 1]) / 255
img = channel(xtrain[n,,], 'rgb')
img[,,1] <- img[,,1]*v[1]
img[,,2] <- img[,,2]*v[2]
img[,,3] <- img[,,3]*v[3]
ff <- t(as.raster(img))
ff[ff == "#000000"] <- "#00000000"
rasterImage(ff, xy1[n,2]-a1, xy1[n,3]-b1,
xy1[n,2]+a1, xy1[n,3]+b1)
}
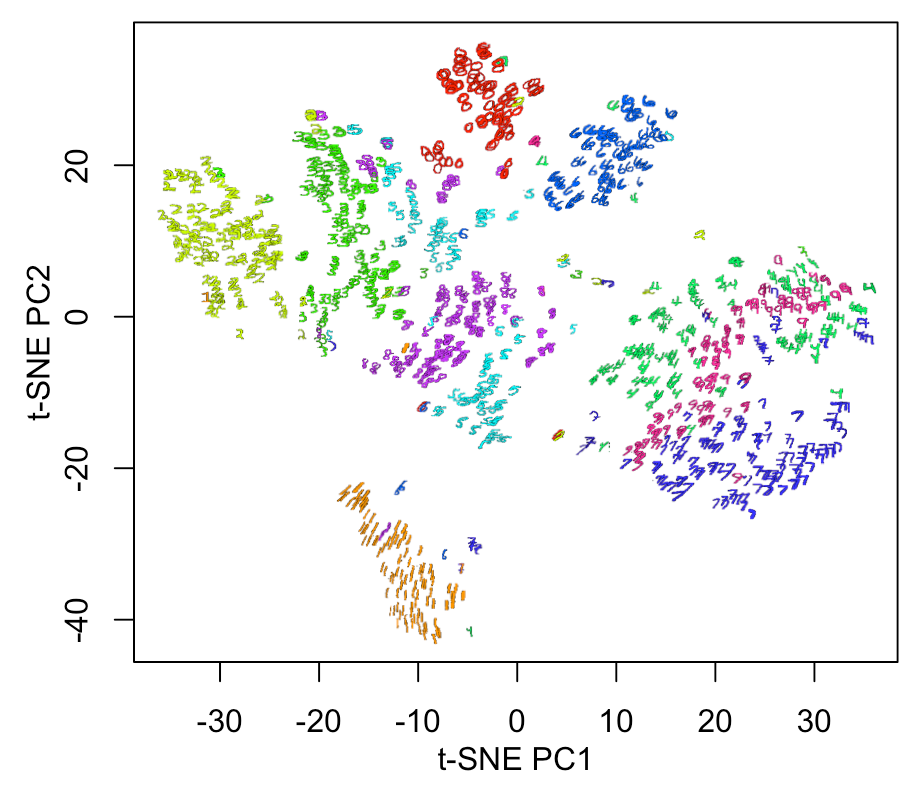
CNNモデルの方*1が、良い感じに分離境界がでています。
色ムラに対するDenoising Autoencoder
このセクションでは、Autoencoderを用いた、色ムラのノイズ除去を行ってみます。
ゴマシオノイズを消す事例は多くあったので、色ムラ戻す事例をやってみます。
まずは、続けてやるとよくないので、RStudioを初期化してみます。
.rs.restartR()
library(keras)
reticulate::use_python("/usr/local/bin/python", required =T)
色ムラがある手書き文字の生成
ランダムな方向に線形の色ムラを施して、4次元アレイ型に変換します。
str(xtrain)
xtrain_noise <- xtrain
abc <- sample(1:4, 1000, replace=T)
for(n in 1:dim(xtrain_noise)[1]){
m <- abc[n]
ab <- matrix(1, 28, 28)*seq(from = 0, to = 1, by = 1/27)
if(m == 1){
xtrain_noise[n,,] <- xtrain[n,,]*ab
}
if(m == 2){
xtrain_noise[n,,] <- xtrain[n,,]*t(ab)
}
if(m == 3){
xtrain_noise[n,,] <- xtrain[n,,]*ab[28:1,]
}
if(m == 4){
xtrain_noise[n,,] <- xtrain[n,,]*t(ab[28:1,])
}
}
x_train <- array_reshape(xtrain, dim=c(dim(xtrain)[1], 28, 28, 1))
x_train_noise <- array_reshape(xtrain_noise, dim=c(dim(xtrain_noise)[1], 28, 28, 1))
そして、色ムラ有無の2D画像を並べて確認してみます。
library(EBImage)
par(mfrow=c(3,2))
for (i in 1:6) {
m <- sample(1:dim(x_train)[1], 1, replace = F)
EBImage::display(combine(t(x_train_noise[m,,,]), t(x_train[m,,,])),
method="raster", nx=2, all=TRUE, spacing = 0.01, margin = 2)
}

左側が色ムラ画像(入力画像)、右側がオリジナル画像(出力画像)を示します。
Autoencoder for denoising モデルの構築
上記モデルとほぼ同じで、filtersは64に変更して構築しました。
input <- layer_input(shape = c(28, 28, 1))
filters <- 64
kernel_size <- c(3,3)
output = input %>%
layer_conv_2d(filters=filters, kernel_size=kernel_size, activation="relu", padding="same") %>%
layer_max_pooling_2d(pool_size=c(2,2), padding="same") %>%
layer_conv_2d(filters=filters, kernel_size=kernel_size, activation="relu", padding="same") %>%
layer_max_pooling_2d(pool_size=c(2,2), padding="same") %>%
layer_conv_2d(filters=filters, kernel_size=kernel_size, activation="relu", padding="same") %>%
layer_upsampling_2d(size=c(2,2)) %>%
layer_conv_2d(filters=filters, kernel_size=kernel_size, activation="relu", padding="same") %>%
layer_upsampling_2d(size=c(2,2)) %>%
layer_conv_2d(filters=1, kernel_size=kernel_size, activation="sigmoid", padding="same")
AutoencoderDenoising <- keras_model(input, output)
summary(AutoencoderDenoising)
モデル表示を行ってみると
source("https://gist.githubusercontent.com/kumeS/41fed511efb45bd55d468d4968b0f157/raw/0f64b83700ac578d0c39abd420da5373d4317083/DL_plot_modi_v1.1.R")
AutoencoderDenoising %>% plot_model_modi(width=1, height=1.25)
(plot_model_modiの出力結果)
tf <- reticulate::import(module = "tensorflow")
py_plot_model <- tf$keras$utils$plot_model
py_plot_model(AutoencoderDenoising, to_file='AutoencoderDenoising_tf.png',
show_shapes=T, show_layer_names=T,
expand_nested=T, dpi=100)
(py_plot_modelの出力結果)
Compileでは、最適化アルゴリズムをadam、損失関数をbinary_crossentropyにした*2。
AutoencoderDenoising %>%
compile(optimizer="adam", loss="binary_crossentropy")
AutoencoderDenoising %>%
fit(x_train_noise, x_train, epochs=200, batch_size=32,
shuffle = T, verbose=1)
このモデルによる色補正の結果を見てみます。
library(EBImage)
pred_imgs <- AutoencoderDenoising %>% predict(x_train_noise)
pred_imgsR <- array_reshape(pred_imgs, dim=c(dim(pred_imgs)[1], 28, 28))
dim(pred_imgsR)
par(mfrow=c(3,2))
for (i in 1:6) {
m <- sample(1:dim(xtrain_noise)[1], 1, replace = F)
EBImage::display(combine(t(xtrain_noise[m,,]), t(pred_imgsR[m,,]), t(xtrain[m,,])),
method="raster", nx=3, all=TRUE, spacing = 0.01, margin = 2)
}

左側が色ムラ画像(入力画像)、真ん中がAutoencoderによるノイズ除去変換後(予測画像)、右側がオリジナル画像(出力画像)です。
トレーニング画像内の内挿であるが、ほぼ色ムラが除去されています。
まとめ
Autoencoderでノイズ除去ができることが分かりました。
また、元画像を入力にして、ノイズ画像を出力にすると、ノイズ付加するAutoencoderモデルとなります。
ただ、実際には、タスクとなるちょうど良いノイズがはいった対応画像を手に入れるのがやや大変そうに思いました。
R/Kerasを用いたDeep Learningの推薦図書


参考文献
www.datatechnotes.com
Autoencoders with Keras, TensorFlow, and Deep Learning - PyImageSearch
elix-tech.github.io













