はじめに
いま。。このブログは、「アニメーショングラフ」推しです。
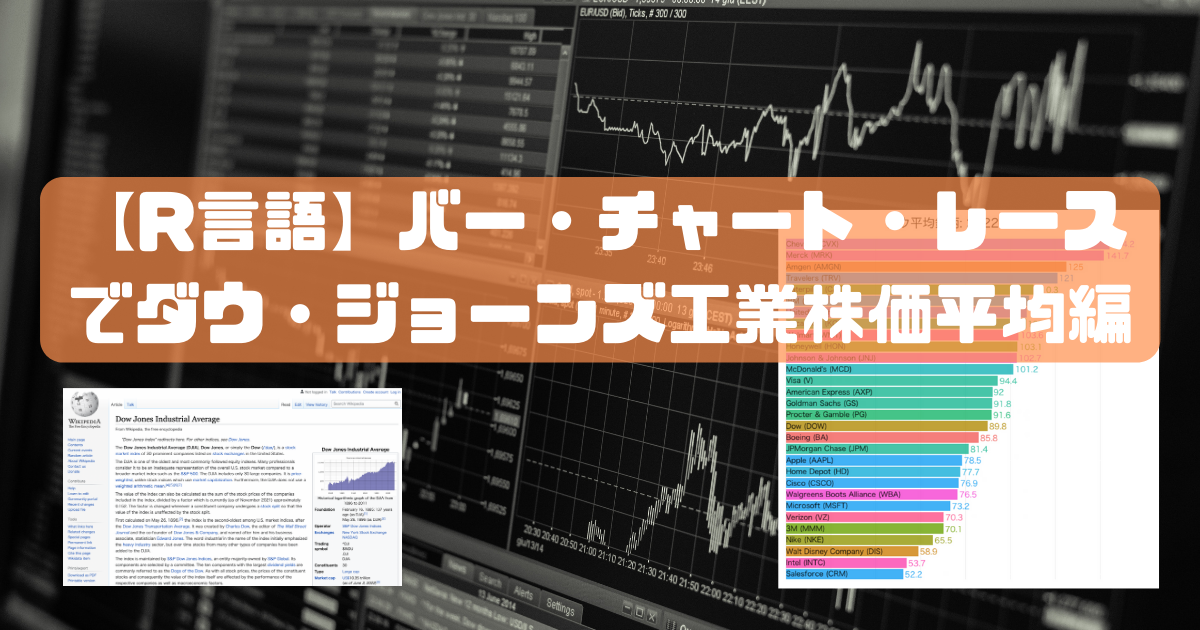
第二弾として、ダウ・ジョーンズ工業株価平均(ダウ平均)の各銘柄の時系列データ(日足)について、「バー・チャート・レース」を作成みました。
ダウ平均は、ちょうど、30銘柄なのでバーチャートにしても見易いです。
さぁ、アニメーショングラフをやっていきましょう。
実行環境
実行環境 macOS Big Sur (バージョン11.5.2) MacBook Air (M1, 2020) チップ Apple M1 メモリ 16GB
Rパッケージの準備
関連のRパッケージを準備します。結構あります。 ここをスキップして、以下のスクリプトを読み込むのでもOKです。
#パッケージ・インストール pack <- c("rvest", "quantmod", "magrittr", "purrr", "tidyr", "ggplot2", "treemapify", "gganimate", "gapminder", "gifski") install.packages(pack[!(pack %in% unique(rownames(installed.packages())))]) #ロード for(n in 1:length(pack)){ eval(parse(text = paste0("library(", pack[n], ")"))) }; rm("n")
getDJIA.Rスクリプトを読み込んでも、Rパッケージがインストールできます。
同時に、今回使用する、getDJIA_list、DJIA_ChartData、DJIA_ChartData_mod、DJIA_animationといった関数群を読み込みます。各関数は、ダウ平均の30銘柄用に調整しています。
#関連関数群の読み込み source("https://gist.githubusercontent.com/kumeS/b4c2a978b31dbe5fc3cb41c4d3e0b6ef/raw/95aefe669ef0ffcccec87e85a87e0d225d73cc71/getDJIA.R")
ではでは、早速、実行してみましょう。
ダウ平均銘柄のデータ取得
まず、getDJIA_list関数では、ウェブスクレイピングを介して、ウィキペディアの該当ページからダウ平均の銘柄リストを取得します。
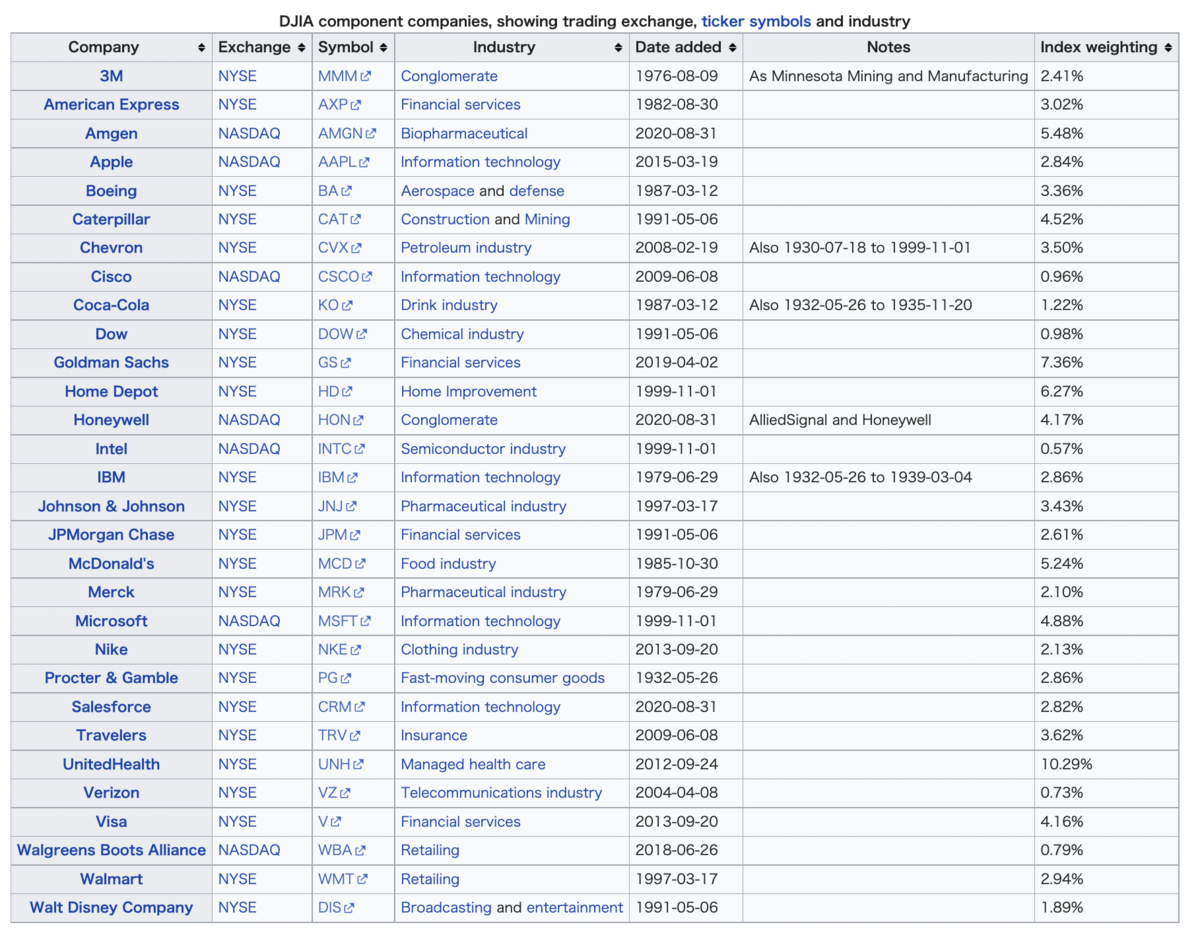
ダウ平均の30銘柄の情報は、この表から取得します。シンボルやセクター情報も含まれます。
それでは、次に、DJIA_ChartData関数でデータを取得します。
#Wikipediaページからダウ平均銘柄リストを取得する DJIAList <- getDJIA_list() #表示 head(DJIAList) # Company Exchange Ticker Sector #1 3M NYSE MMM Conglomerate #2 American Express NYSE AXP Financial services #3 Amgen NASDAQ AMGN Biopharmaceutical #4 Apple NASDAQ AAPL Information technology #5 Boeing NYSE BA Aerospace and defense #6 Caterpillar NYSE CAT Construction and Mining #銘柄のセクター数 table(DJIAList$Sector) #ダウ平均のデータ取得: 実行時間 2分くらい ChartData <- DJIA_ChartData(Dat=DJIAList, term=c("2022-01-01", "2022-12-31")) #いったん、RDS保存 saveRDS(ChartData, "DJIA_ChartData.Rds") #ChartData <- readRDS("DJIA_ChartData.Rds") #または、DJIA_ChartData.Rdsをダウンロードできるようにもしています。 #system("which svn") #system("svn export https://github.com/kumeS/Blog/trunk/R_BarChart/DJIA_ChartData.Rds") #表示 head(ChartData) # Company Ticker Sector date close dclose dclose2 ranking #1 3M MMM Conglomerate 2022-01-03 100 0 #F0F5F0 1 #2 American Express AXP Financial services 2022-01-03 100 0 #F0F5F0 2 #3 Amgen AMGN Biopharmaceutical 2022-01-03 100 0 #F0F5F0 3 #4 Apple AAPL Information technology 2022-01-03 100 0 #F0F5F0 4 #5 Boeing BA Aerospace and defense 2022-01-03 100 0 #F0F5F0 5 #6 Caterpillar CAT Construction and Mining 2022-01-03 100 0 #F0F5F0 6
このステップで、ダウ平均の30銘柄のリストとデータが取得できました。
ダウ平均銘柄でのバー・チャート・レース
ダウ平均銘柄のバー・チャート・レース作成
次に、ダウ平均銘柄のデータを使って、バー・チャート・レースを作成します。
DJIA_ChartData_mod関数で、ダウ平均の全銘柄のデータフレームにします。この関数で、rankingという列データを作成します。その後、DJIA_animation関数を実行して、チャート設定をします。また、日本語設定も別途します。
#データ補正: 全銘柄を取得する ChartData1 <- DJIA_ChartData_mod(ChartData) #表示 head(ChartData1) # Company Ticker Sector CompanyTicker CompanySector date #1 3M MMM Conglomerate 3M (MMM) 3M (Conglomerate) 2022-01-03 #31 3M MMM Conglomerate 3M (MMM) 3M (Conglomerate) 2022-01-04 #61 3M MMM Conglomerate 3M (MMM) 3M (Conglomerate) 2022-01-05 #91 3M MMM Conglomerate 3M (MMM) 3M (Conglomerate) 2022-01-06 #121 3M MMM Conglomerate 3M (MMM) 3M (Conglomerate) 2022-01-07 #151 3M MMM Conglomerate 3M (MMM) 3M (Conglomerate) 2022-01-10 # close dclose dclose2 ranking #1 100.0 0.0 #F0F5F0 1 #31 101.4 1.4 #F0F5F0 12 #61 101.0 1.0 #F0F5F0 15 #91 100.1 0.1 #F0F5F0 15 #121 101.2 1.2 #F0F5F0 14 #日本語設定: 必須 ggplot2::update_geom_defaults("text", list(family = "HiraKakuPro-W3")) ggplot2::update_geom_defaults("label", list(family = "HiraKakuPro-W3")) ##実行 p1 <- DJIA_animation(ChartData1, Label="ダウ平均銘柄") #gif出力: 実行時間 30秒くらい animate(p1, fps = 2, duration = 125, width = 700, height = 700, renderer = gifski_renderer("DJIA_BarChartRace_animation_p1.gif"))

まとめ
今回、ダウ平均銘柄の2022年12月まで値動きのバー・チャート・レースを作成しました。 全体を眺めてみると、12月20日現在で、2022年年初からで100%以上なっている銘柄は、約10銘柄あるみたいですね。 そのなかでも、シェブロン(CVX)とメルク(MRK)が結構上昇していて、目立ちますね。2022年は、石油と医薬品の年でしたね。