- はじめに
- Keras/TensorFlowのセットアップ
- 手書き文字MNISTデータセットの用意
- Autoencoderのモデル構築
- まとめ
- R/Kerasを用いたDeep Learningの推薦図書
- 全コード in Gist
- 補足
はじめに
Rで、ディープラーニング( Deep Learning )をやるというのが最近の活動である。
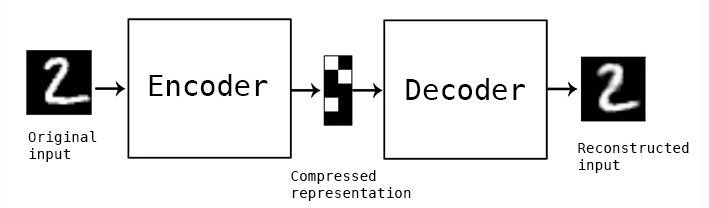
【1】では、教師なしニューラルネットワークであるAutoencoder(オートエンコーダー)のsimple modelを実装してみる。
Autoencoder は、Encoder(元データから低次元への変換層)とDecoder(低次元から元データに戻す変換層)からなるニューラルネットワークの特殊系の1つである。 Autoencoderでは、出力層で与える教師データを入力層としても与えることで、 入力層と出力層のユニットが同じとなる構造を持つ。
Autoencoderの開発は1980年代に遡り、Hinton と PDPグループが、 「教師なしの逆伝播」の問題に対処するために、教師データを入力データとして使用したことに始まる*1*2。
また、2006年、Hinton と Salakhutdinov は、多層のニューラルネットワークを持つDeep Autoencoderを用いた、手書きアラビア数字画像などの次元圧縮の結果を報告した*3。
彼らは、小さな中央層を持つ多層ニューラルネットワークをトレーニングさせて、高次元の入力ベクトルを出力層で再構築することで、画像データを低次元のコードとして変換できることを示した。また、データの次元数を削減するツールとして、主成分分析よりも、Deep autoencoderのほうが優れていると述べている。
このとき、両者とも画像をいったんベクトルデータに変換しているが、 確かに、PCA(下図の左)よりもだいぶ良く見える結果である。

*4の論文より引用
実際に、2次元上に数字画像をプロットした結果がサプリメント図にのっている。

*5のサプリメントデータより引用
今回、R言語のKeras/TensorFlowパッケージを使って、Autoencoderのmodelをいろいろと実装していく。そして、サプリ図のような、数字画像のプロット結果を得ることをゴールにしている。
Rでの、Keras/TensorFlowのインストールについては以下を参照のこと。
www.slideshare.net
Keras/TensorFlowのセットアップ
今回、CNN(Convolutional Neural Network: 畳み込みニューラルネットワーク)は使わないので、AutoencoderはCPU実行した。
# Kerasパッケージのロード library(keras) # "/usr/local/bin/python"の選択 reticulate::use_python("/usr/local/bin/python", required =T)
ここで、「install_keras()」は特にやらなくても動くはずである。
BrewでインストールしたPythonを使っていて、Pythonのパスは以下のように「/usr/local/bin/python」になっている。
reticulate::py_config() python: /usr/local/bin/python libpython: /usr/local/opt/python/Frameworks/Python.framework/Versions/3.7/lib/python3.7/config-3.7m-darwin/libpython3.7.dylib pythonhome: /usr/local/Cellar/python/3.7.7/Frameworks/Python.framework/Versions/3.7:/usr/local/Cellar/python/3.7.7/Frameworks/Python.framework/Versions/3.7 version: 3.7.7 (default, Mar 10 2020, 15:43:33) [Clang 11.0.0 (clang-1100.0.33.17)] numpy: /Users/skume/Library/Python/3.7/lib/python/site-packages/numpy numpy_version: 1.16.3 tensorflow: /usr/local/lib/python3.7/site-packages/tensorflow NOTE: Python version was forced by use_python function
このあたりの設定についは、過去のブログ記事を参照のこと。
https://skume.hatenablog.com/entry/2020/05/10/225341skume.hatenablog.com
手書き文字MNISTデータセットの用意
Autoencoderは、以下の図のように、中央に圧縮特徴層を持ち、その前後にエンコード層とデコード層の組み合わせとして表される*6。
まずは、あの有名な手書き文字のデータセットをダウンロードしてアレイ型に変換する。
#MNISTのダウンロード Data <- dataset_mnist() str(Data) #アレイ型に変換 xtrain <- Data$train$x xtrain <- xtrain/255 ytrain <- Data$train$y xtest <- Data$test$x xtest <- xtest/255 ytest <- Data$test$y
今回、2D画像は一次元ベクトルに変換して入力データとするため inputユニットサイズ = ピクセル数となり、それを計算しておく。
# インプットデータサイズの計算 dim(xtrain) input_size <- dim(xtrain)[2]*dim(xtrain)[3] input_size
inputサイズは、28 x 28 = 784ユニットとなる。
さらに、3次元アレイ(1: 画像、2: X、3: Y)から、2次元アレイ(1: 画像、2: 一次元ベクトル)に変換する。
dim(xtrain) #[1] 60000 28 28 dim(xtest) #[1] 10000 28 28 #2次元アレイに変換 x_train = array_reshape(xtrain, dim=c(dim(xtrain)[1], input_size)) x_test = array_reshape(xtest, dim=c(dim(xtest)[1], input_size)) dim(x_train) #[1] 60000 784 dim(x_test) #[1] 10000 784
Autoencoderのモデル構築
ここから、実際のAutoencoderのモデル構築を行う。
Rでは、パイプ文法(%>%)でモデルを書いていく。
シンプルAutoencoderモデル 01
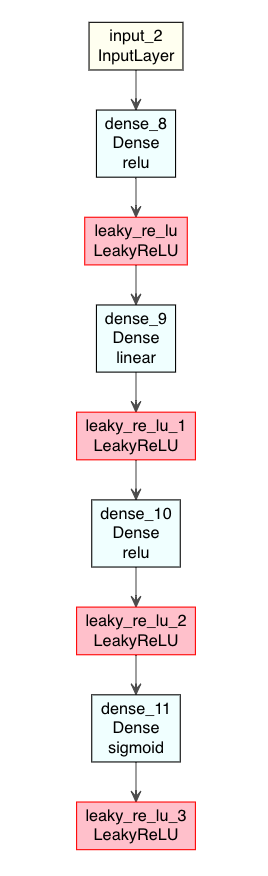
input1 <- layer_input(shape = input_size) output1 <- input1 %>% layer_dense(units=256, activation = "relu") %>% layer_activation_leaky_relu() %>% layer_dense(units=2) %>% layer_activation_leaky_relu() %>% layer_dense(units=256, activation = "relu") %>% layer_activation_leaky_relu() %>% layer_dense(units = input_size, activation = "sigmoid") %>% layer_activation_leaky_relu() # モデル構築 Autoencoder1 <- keras_model(input1, output1)
モデルをsummaryで表示してみる。
summary(Autoencoder1) #Model: "model" #___________________________________________________________ #Layer (type) Output Shape Param # #=========================================================== #input_1 (InputLayer) [(None, 784)] 0 #___________________________________________________________ #dense (Dense) (None, 256) 200960 #___________________________________________________________ #leaky_re_lu (LeakyReLU) (None, 256) 0 #___________________________________________________________ #dense_1 (Dense) (None, 2) 514 #___________________________________________________________ #leaky_re_lu_1 (LeakyReLU) (None, 2) 0 #___________________________________________________________ #dense_2 (Dense) (None, 256) 768 #___________________________________________________________ #leaky_re_lu_2 (LeakyReLU) (None, 256) 0 #___________________________________________________________ #dense_3 (Dense) (None, 784) 201488 #___________________________________________________________ #leaky_re_lu_3 (LeakyReLU) (None, 784) 0 #=========================================================== #Total params: 403,730 #Trainable params: 403,730 #Non-trainable params: 0 #___________________________________________________________
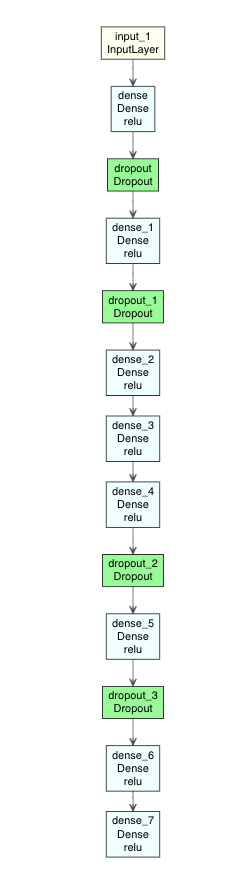
DLのモデルはグラフ構造の方が見やすいので、モデルの図示を試してみる。
Rでは、deepvizパッケージ*7で、ニューラルネットワーク構造を可視化できる。
ただ、deepvizは色合いが短調なので、ここでは改変版のplot_model_modiで行う。
# Gistで公開している plot_model_modi のロード source("https://gist.githubusercontent.com/kumeS/41fed511efb45bd55d468d4968b0f157/raw/07da3ba4a2e477f352d03e8b5ac00d394fe9afec/DL_plot_modi_v1.1.R") #Autoencoder1モデルの図示 modelplot <- Autoencoder1 modelplot %>% plot_model_modi()
(plot_model_modiの出力結果)
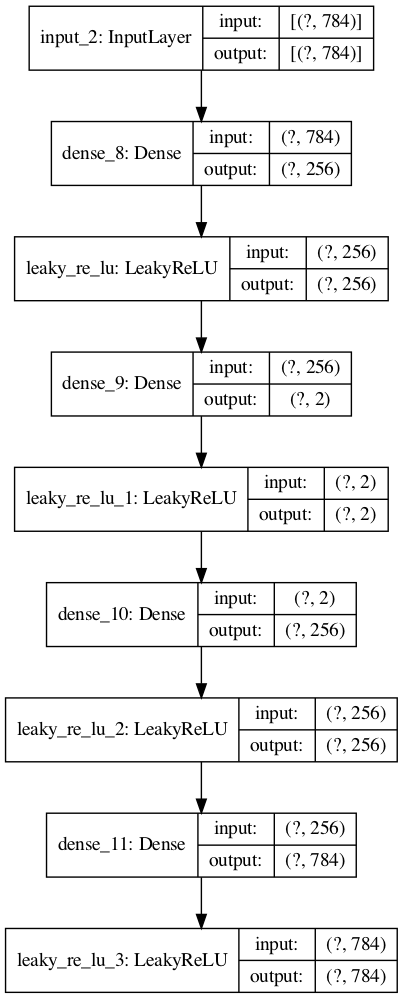
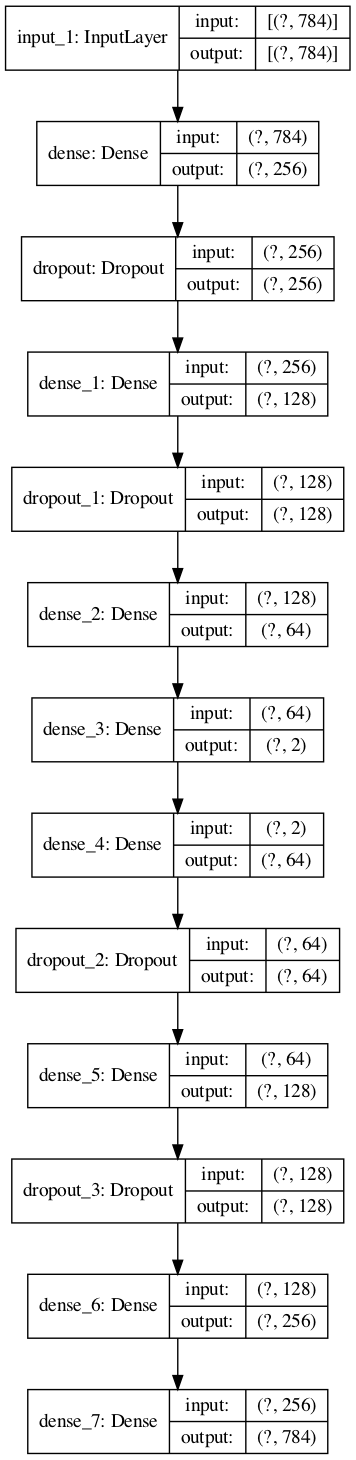
また、Python TensorFlowでのplot_modelを行いたい場合には、
R上に、PythonのTensorFlowパッケージをロードすれば、グラフ出力ができる。
#Python Tensorflowのplot_modelを使う tf <- reticulate::import(module = "tensorflow") py_plot_model <- tf$keras$utils$plot_model py_plot_model(modelplot, to_file='model1.png', show_shapes=T, show_layer_names=T, expand_nested=T, dpi=100)
(Python Tensorflowのplot_modelの出力結果)
次に、モデルのコンパイルを行う。
最適化アルゴリズムはrmspropのデフォルト値、
損失関数はmean_squared_errorにしている。
# Compile Autoencoder1 %>% compile(optimizer="rmsprop", loss="mean_squared_error")
そして、フィット でAutoencoderを実行する。
# Fit Autoencoder1 %>% fit(x_train, x_train, epochs=100, batch_size=1000)
次に、AutoencoderによるTrainデータセットの変換結果を確認する。
まずは、アレイ型を2次元から3次元に変換する。
pred_imgs1 <- Autoencoder1 %>% predict(x_train) pred_imgsR1 <- array_reshape(pred_imgs1, dim=c(dim(pred_imgs1)[1], 28, 28)) dim(pred_imgsR1) #[1] 60000 28 28 #Testデータセットについて #(今回はスキップ) #pred_imgsY1 = Autoencoder %>% predict(x_test) #pred_imgsRY1 = array_reshape(pred_imgsY1, dim=c(dim(pred_imgsY1)[1], 28, 28))
手書き文字を表示するのに、まずは、 BioconductorのEBImage*8をインストールする。
if (!requireNamespace("BiocManager", quietly = TRUE)){install.packages("BiocManager")} BiocManager::install("EBImage") #EBImageをロードする library(EBImage)
input画像とAutoencoderをかけた後の画像の結果を並べて表示してみる。
par(mfrow=c(3,2)) for (i in 1:6) { m <- sample(1:dim(xtrain)[1], 1, replace = F) display(combine(t(xtrain[m,,]), t(pred_imgsR1[m,,])), method="raster", nx=2, all=TRUE, spacing = 0.01, margin = 2) }
入力(左側)と出力(右側)の結果を示す。
やはり論文のサプリ図のように、圧縮特徴層を取り出して、文字画像データを図上にプロットしたくなる。
まずは、中間層を抜き出して、2次元プロットを試みる。
#Autoencoderの中間層までのモデルを抜き出す intermediate_layer <- keras_model(inputs = Autoencoder1$input, outputs = get_layer(Autoencoder1, "dense_1")$output) summary(intermediate_layer) #Model: "model_1" #____________________________________________________________________ #Layer (type) Output Shape Param # #==================================================================== #input_1 (InputLayer) [(None, 784)] 0 #____________________________________________________________________ #dense (Dense) (None, 256) 200960 #____________________________________________________________________ #leaky_re_lu (LeakyReLU) (None, 256) 0 #____________________________________________________________________ #dense_1 (Dense) (None, 2) 514 #==================================================================== #Total params: 201,474 #Trainable params: 201,474 #Non-trainable params: 0 #____________________________________________________________________ #さらに、中間層まで変換をpredictする intermediate_output <- predict(intermediate_layer, x_train)
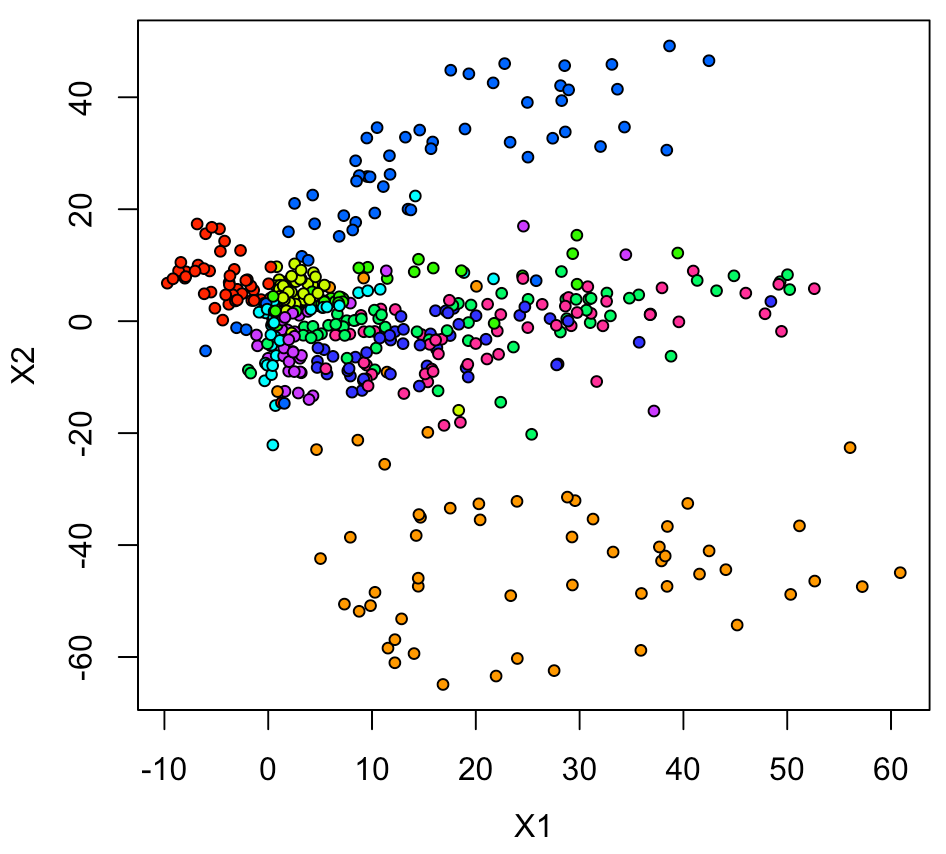
とりあえず、色付きマーカーで2Dプロットをしてみる。
全てのデータ(6万点)だと多過ぎるので、500点分をランダムサンプリングして表示した。
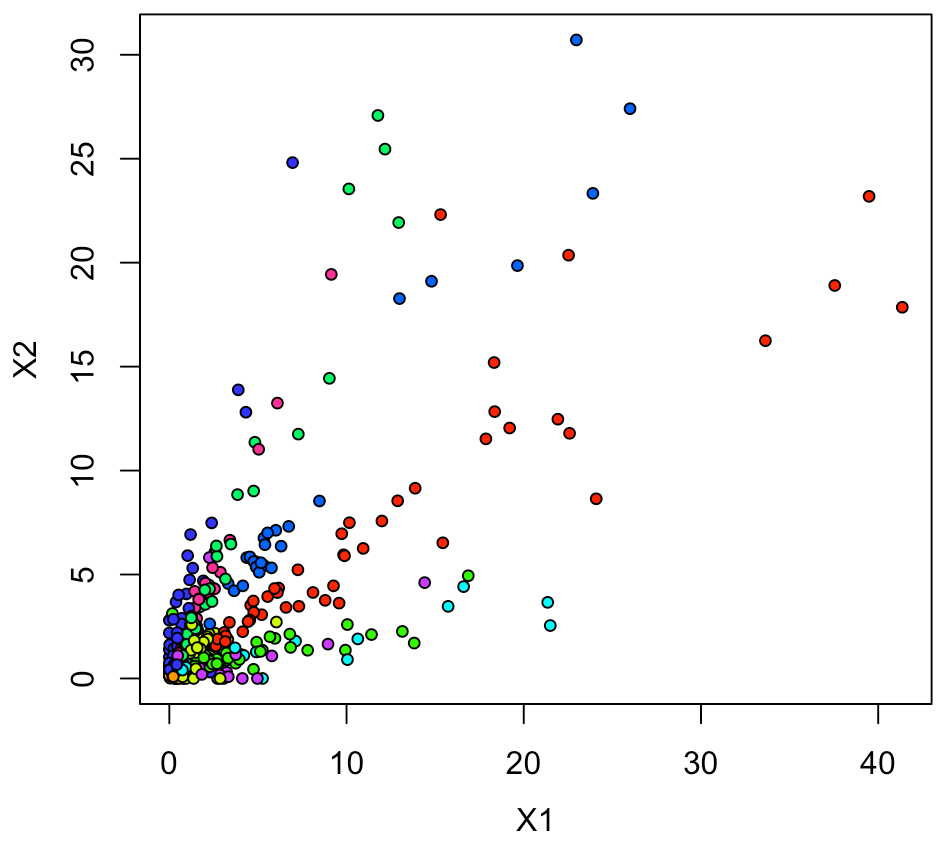
xy <- data.frame(ytrain, intermediate_output) Sam <- sample(1:nrow(xy), 500, replace = F) xy1 <- xy[Sam,] par(mfrow=c(1,1), mai=c(0.75,0.75,0.2,0.2), mgp = c(2.5,1,0)) plot(xy1[,2:3], pch=21, cex=0.75, bg=rainbow(10)[xy1[,1]+1])
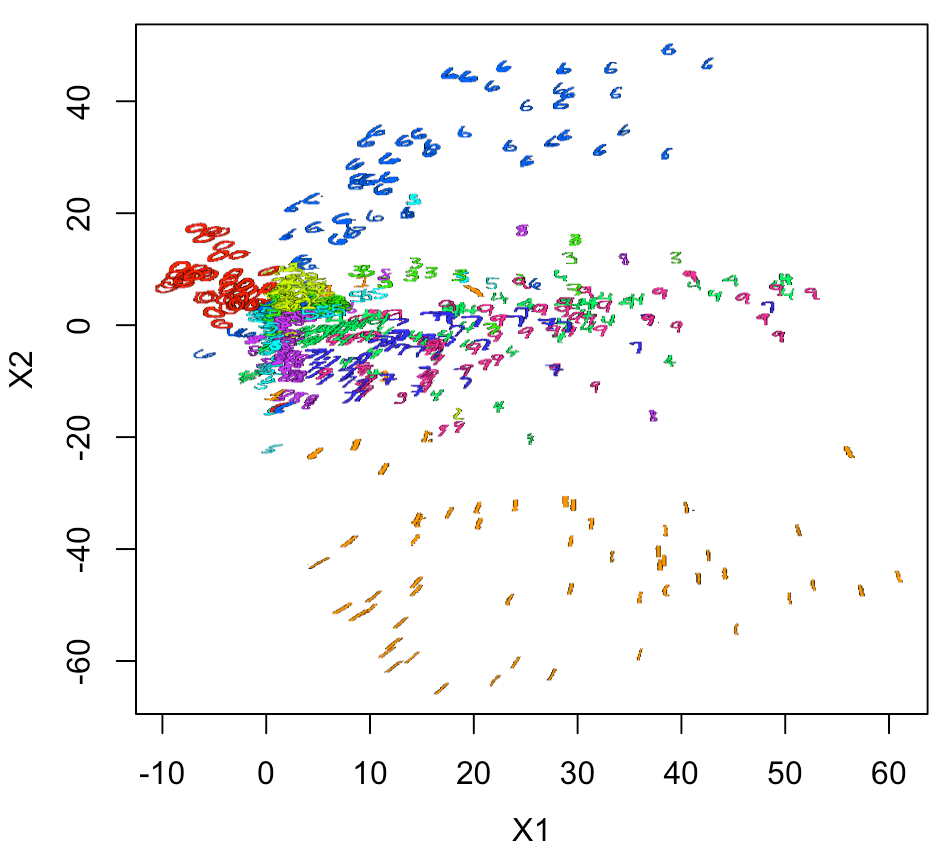
やはり、実際の数字画像でプロットしないと雰囲気が出ない。
ちょっと面倒なのだが、空白のプロットをして、 そこにラスター画像を重ねていくとできる。 ラスター画像は背景を透明にしているのがひと工夫である。
#まずは、空白のプロットをしてみる plot(xy1[,2:3], pch=21, cex=0.1, col="white") ##そこに、画像を色付きでプロットする for(n in 1:nrow(xy1)){ #n <-4 v <- col2rgb(rainbow(10)[xy1[n,1] + 1]) / 255 img = channel(xy2[n,,], 'rgb') img[,,1] <- img[,,1]*v[1] img[,,2] <- img[,,2]*v[2] img[,,3] <- img[,,3]*v[3] ff <- t(as.raster(img)) ff[ff == "#000000"] <- "#00000000" rasterImage(ff, xy1[n,2]-1.5, xy1[n,3]-1.5, xy1[n,2]+1.5, xy1[n,3]+1.5) }
プロット結果として考察できることは、 このモデルは、0、1、6以外ではあまり精度が良くない。
そこで、別モデルもいくつか試してみる。
RStudioセッションの初期化
レイヤーが連番となるため、DL実行ごとに、いったん、Rセッションを初期化しておくのが無難・・・
.rs.restartR() library(keras) library(EBImage) reticulate::use_python("/usr/local/bin/python", required =T)
シンプルAutoencoderモデル 02
このモデルでは、少しレイヤー数を増やして、Dropoutも入れてみた。
活性化関数は、reluにしている。
input2 <- layer_input(shape = input_size) output2 <- input2 %>% layer_dense(units = 256, activation = "relu") %>% layer_dropout(rate = 0.2) %>% layer_dense(units = 128, activation = "relu") %>% layer_dropout(rate = 0.1) %>% layer_dense(units = 64, activation = "relu") %>% layer_dense(units = 2, activation = "relu") %>% layer_dense(units = 64, activation = "relu") %>% layer_dropout(rate = 0.1) %>% layer_dense(units = 128, activation = "relu") %>% layer_dropout(rate = 0.2) %>% layer_dense(units = 256, activation = "relu") %>% layer_dense(units = input_size, activation = "relu") Autoencoder2 <- keras_model(input2, output2) summary(Autoencoder2)
モデルをプロットするとこんな感じである。
modelplot <- Autoencoder2 modelplot %>% plot_model_modi()
(plot_model_modiの出力結果)
tf <- reticulate::import(module = "tensorflow") py_plot_model <- tf$keras$utils$plot_model py_plot_model(modelplot, to_file='model.png', show_shapes=T, show_layer_names=T, expand_nested=T, dpi=100)
(Python Tensorflowのplot_modelの出力結果)
次に、コンパイル・フィットを実行する。
最適化アルゴリズムはrmspropのデフォルト値、
損失関数はmean_squared_errorにしている。
#compile Autoencoder2 %>% compile(optimizer="rmsprop", loss="mean_squared_error") #Fit Autoencoder2 %>% fit(x_train, x_train, epochs=100, batch_size=1000, shuffle=TRUE)
(計算結果)
AutoencoderによるTrainデータセットの変換結果を確認してみると、
pred_imgs2 <- Autoencoder2 %>% predict(x_train) pred_imgsR2 <- array_reshape(pred_imgs2, dim=c(dim(pred_imgs2)[1], 28, 28)) dim(pred_imgsR2) par(mfrow=c(3,2)) for (i in 1:6) { m <- sample(1:dim(xtrain)[1], 1, replace = F) display(combine(t(xtrain[m,,]), t(pred_imgsR2[m,,])), method="raster", nx=2, all=TRUE, spacing = 0.01, margin = 2) }
入力(左側)と出力(右側)の結果を示す。
同じく、中間層の圧縮特徴量でプロットしてみる。
##中間層の取得 summary(Autoencoder2) intermediate_layer <- keras_model(inputs = Autoencoder2$input, outputs = get_layer(Autoencoder2, "dense_3")$output) summary(intermediate_layer) intermediate_output <- predict(intermediate_layer, x_train) #2Dプロット xy <- data.frame(ytrain, intermediate_output) Sam <- sample(1:nrow(xy), 500, replace = F) xy1 <- xy[Sam,] par(mfrow=c(1,1), mai=c(0.75,0.75,0.2,0.2), mgp = c(2.5,1,0)) plot(xy1[,2:3], pch=21, cex=0.75, bg=rainbow(10)[xy1[,1]+1])
これでは、データ点がゼロ付近に密集している印象なので、 XY軸それぞれでlog10をとることで、それが解消できるか試してみる。
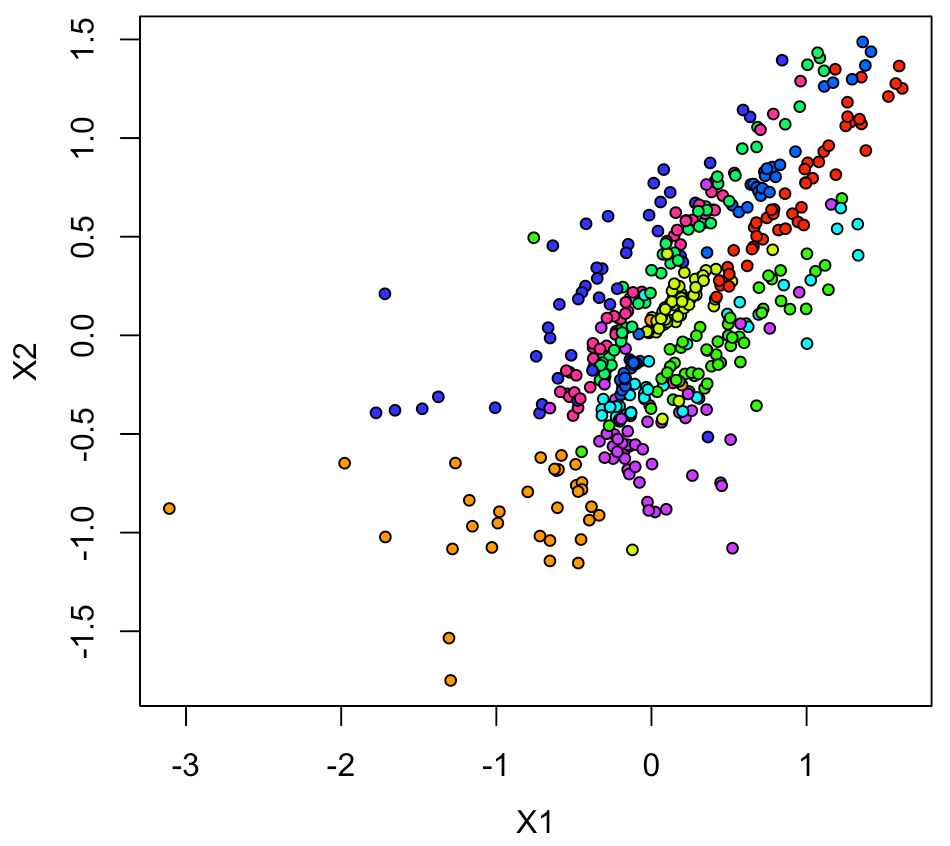
そうすると、結構いい感じでプロットされた。
xy1[,2:3] <- log10(xy1[,2:3]) plot(xy1[,2:3], pch=21, cex=0.75, bg=rainbow(10)[xy1[,1]+1])
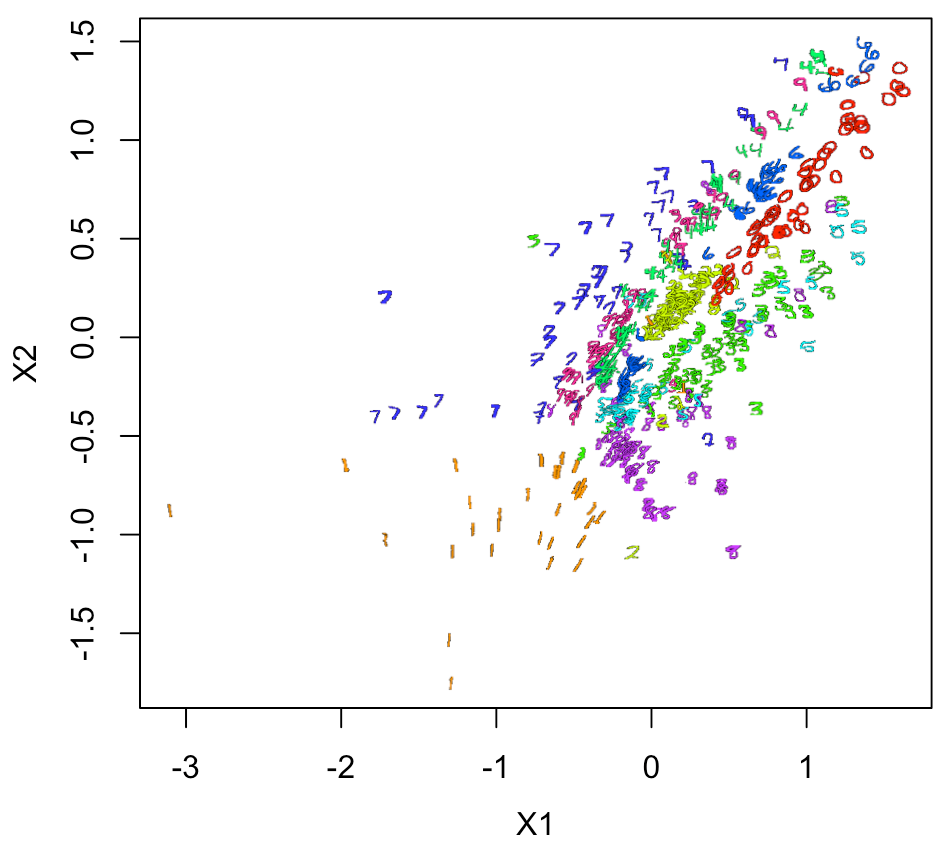
次に、Log10をとったXY座標で、実際の画像での2Dプロットをやってみると
xy2 <- xtrain[Sam,,] #空白のプロット plot(xy1[,2:3], pch=21, cex=0.1, col="white") ##プロット全体のXY座標サイズを考慮して、全体の3%の大きさでプロットする a <- range(xy1[,2][is.finite(xy1[,2])]) b <- range(xy1[,3][is.finite(xy1[,3])]) a1 <- diff(a)*0.015 b1 <- diff(b)*0.015 ##色付き画像でプロットする for(n in 1:nrow(xy1)){ #n <-4 v <- col2rgb(rainbow(10)[xy1[n,1] + 1]) / 255 img = channel(xy2[n,,], 'rgb') img[,,1] <- img[,,1]*v[1] img[,,2] <- img[,,2]*v[2] img[,,3] <- img[,,3]*v[3] ff <- t(as.raster(img)) ff[ff == "#000000"] <- "#00000000" rasterImage(ff, xy1[n,2]-a1, xy1[n,3]-b1, xy1[n,2]+a1, xy1[n,3]+b1) }
結構良い感じにできる。
シンプルAutoencoderモデル 03
最後に、論文あった、レイヤーのユニット数(784-1000-500-250-2)で実装してみる。
3回目なので、説明はやや端折り気味で。。
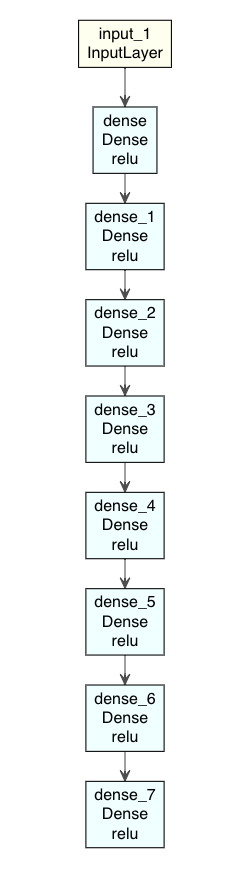
input3 <- layer_input(shape = input_size) output3 <- input3 %>% layer_dense(units = 1000, activation = "relu") %>% layer_dense(units = 500, activation = "relu") %>% layer_dense(units = 250, activation = "relu") %>% layer_dense(units = 2, activation = "relu") %>% layer_dense(units = 250, activation = "relu") %>% layer_dense(units = 500, activation = "relu") %>% layer_dense(units = 100, activation = "relu") %>% layer_dense(units = input_size, activation = "relu") Autoencoder3 <- keras_model(input3, output3) summary(Autoencoder3) #Model: "model" #______________________________________________________________________ #Layer (type) Output Shape Param # #====================================================================== #input_1 (InputLayer) [(None, 784)] 0 #______________________________________________________________________ #dense (Dense) (None, 1000) 785000 #______________________________________________________________________ #dense_1 (Dense) (None, 500) 500500 #______________________________________________________________________ #dense_2 (Dense) (None, 250) 125250 #______________________________________________________________________ #dense_3 (Dense) (None, 2) 502 #______________________________________________________________________ #dense_4 (Dense) (None, 250) 750 #______________________________________________________________________ #dense_5 (Dense) (None, 500) 125500 #______________________________________________________________________ #dense_6 (Dense) (None, 100) 50100 #______________________________________________________________________ #dense_7 (Dense) (None, 784) 79184 #====================================================================== #Total params: 1,666,786 #Trainable params: 1,666,786 #Non-trainable params: 0 #______________________________________________________________________
モデルを図示すると、
modelplot <- Autoencoder3 modelplot %>% plot_model_modi(width=1, height=1.25)
(plot_model_modiの出力結果)
続けて、コンパイルして、フィットを実行する。
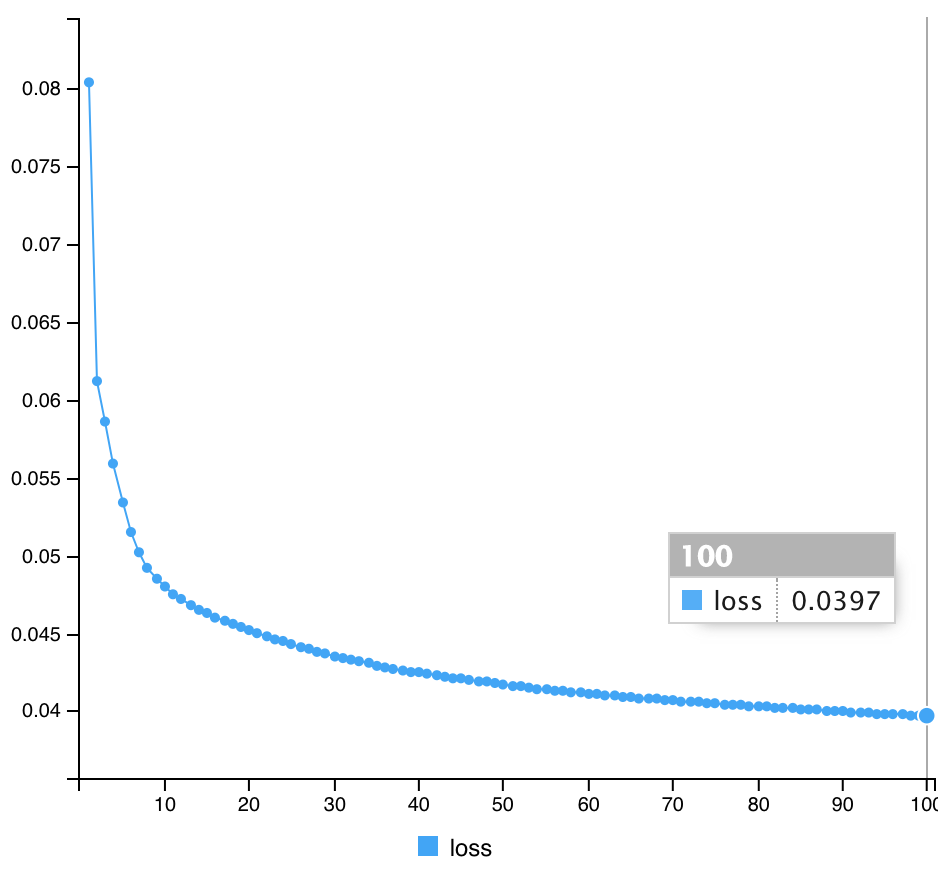
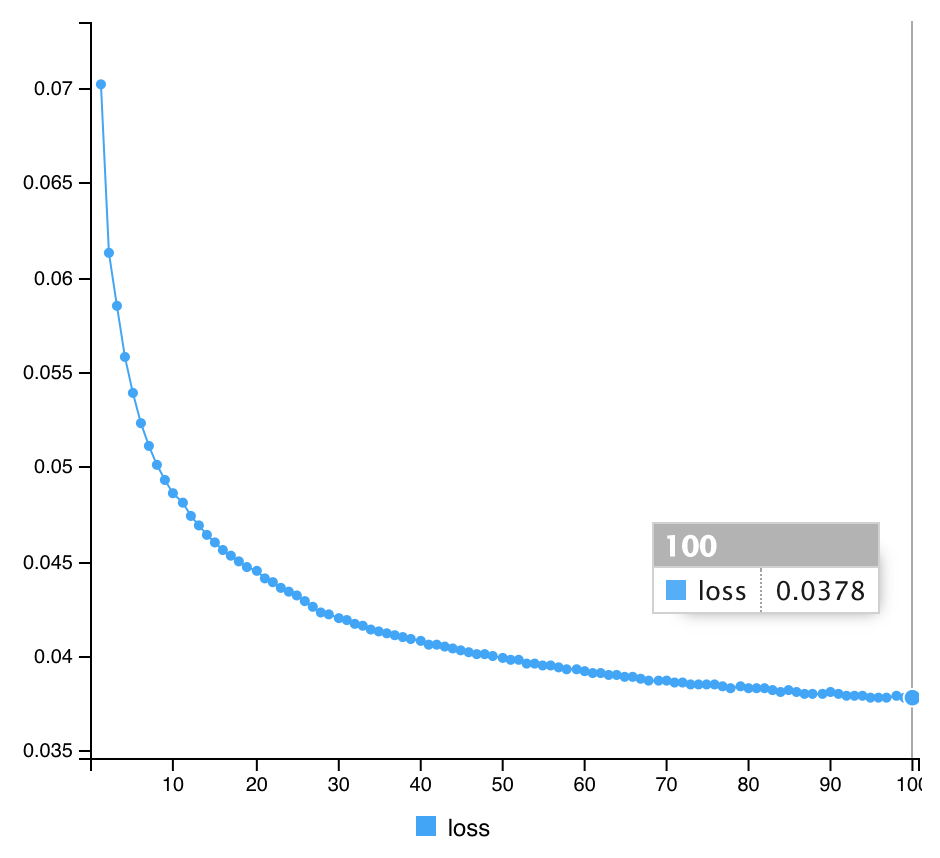
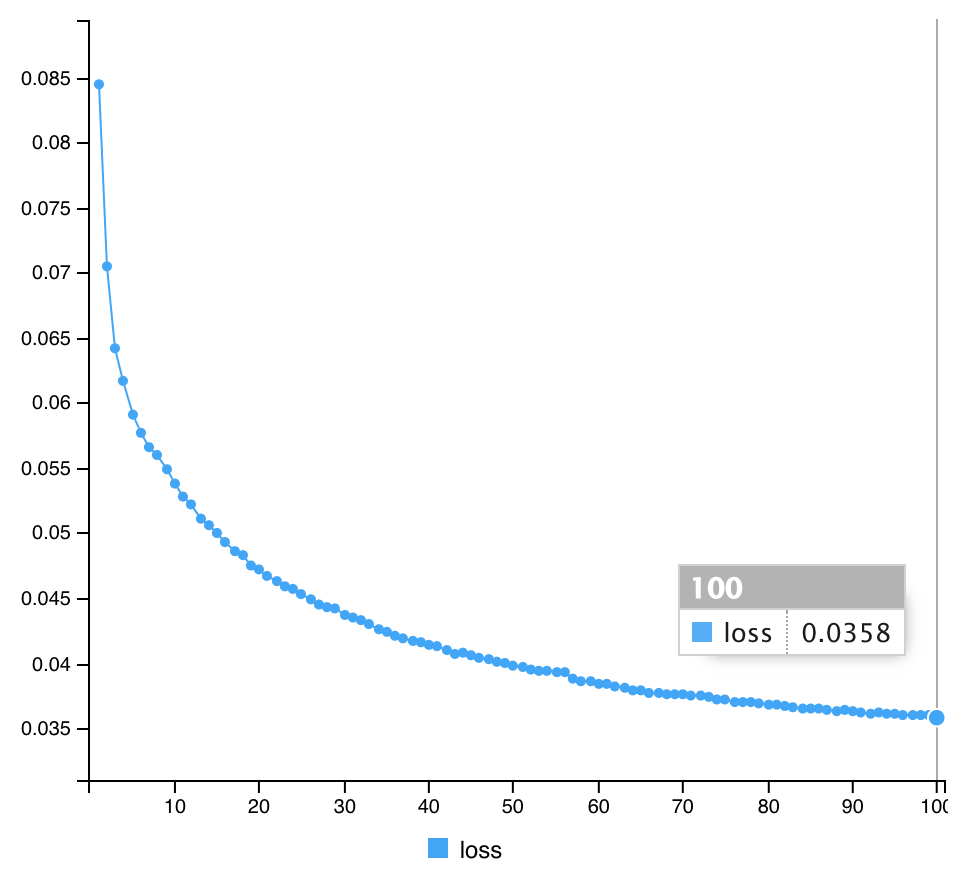
#compile Autoencoder3 %>% compile(optimizer="rmsprop", loss="mean_squared_error") #Fit Autoencoder3 %>% fit(x_train, x_train, epochs=100, batch_size=1000, shuffle=TRUE) #計算途中の出力 Epoch 1/100 60/60 [==============================] - 3s 48ms/step - loss: 0.0845 Epoch 2/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0705 Epoch 3/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0642 Epoch 4/100 60/60 [==============================] - 3s 52ms/step - loss: 0.0617 Epoch 5/100 60/60 [==============================] - 3s 46ms/step - loss: 0.0591 Epoch 6/100 60/60 [==============================] - 3s 44ms/step - loss: 0.0577 Epoch 7/100 60/60 [==============================] - 3s 42ms/step - loss: 0.0566 Epoch 8/100 60/60 [==============================] - 2s 42ms/step - loss: 0.0560 Epoch 9/100 60/60 [==============================] - 3s 42ms/step - loss: 0.0549 Epoch 10/100 60/60 [==============================] - 3s 47ms/step - loss: 0.0538 Epoch 11/100 60/60 [==============================] - 3s 47ms/step - loss: 0.0528 Epoch 12/100 60/60 [==============================] - 3s 47ms/step - loss: 0.0522 Epoch 13/100 60/60 [==============================] - 3s 46ms/step - loss: 0.0511 Epoch 14/100 60/60 [==============================] - 3s 44ms/step - loss: 0.0506 Epoch 15/100 60/60 [==============================] - 3s 42ms/step - loss: 0.0500 Epoch 16/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0493 Epoch 17/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0486 Epoch 18/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0483 Epoch 19/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0475 Epoch 20/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0472 Epoch 21/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0467 Epoch 22/100 60/60 [==============================] - 3s 42ms/step - loss: 0.0463 Epoch 23/100 60/60 [==============================] - 3s 42ms/step - loss: 0.0459 Epoch 24/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0457 Epoch 25/100 60/60 [==============================] - 3s 44ms/step - loss: 0.0453 Epoch 26/100 60/60 [==============================] - 3s 42ms/step - loss: 0.0449 Epoch 27/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0445 Epoch 28/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0443 Epoch 29/100 60/60 [==============================] - 3s 45ms/step - loss: 0.0442 Epoch 30/100 60/60 [==============================] - 3s 44ms/step - loss: 0.0437 Epoch 31/100 60/60 [==============================] - 3s 49ms/step - loss: 0.0435 Epoch 32/100 60/60 [==============================] - 4s 62ms/step - loss: 0.0433 Epoch 33/100 60/60 [==============================] - 5s 79ms/step - loss: 0.0430 Epoch 34/100 60/60 [==============================] - 4s 65ms/step - loss: 0.0426 Epoch 35/100 60/60 [==============================] - 3s 48ms/step - loss: 0.0424 Epoch 36/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0421 Epoch 37/100 60/60 [==============================] - 3s 42ms/step - loss: 0.0419 Epoch 38/100 60/60 [==============================] - 3s 45ms/step - loss: 0.0417 Epoch 39/100 60/60 [==============================] - 3s 44ms/step - loss: 0.0416 Epoch 40/100 60/60 [==============================] - 3s 45ms/step - loss: 0.0414 Epoch 41/100 60/60 [==============================] - 3s 47ms/step - loss: 0.0413 Epoch 42/100 60/60 [==============================] - 3s 46ms/step - loss: 0.0410 Epoch 43/100 60/60 [==============================] - 3s 46ms/step - loss: 0.0407 Epoch 44/100 60/60 [==============================] - 3s 47ms/step - loss: 0.0408 Epoch 45/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0406 Epoch 46/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0404 Epoch 47/100 60/60 [==============================] - 3s 44ms/step - loss: 0.0403 Epoch 48/100 60/60 [==============================] - 3s 46ms/step - loss: 0.0401 Epoch 49/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0400 Epoch 50/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0398 Epoch 51/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0397 Epoch 52/100 60/60 [==============================] - 3s 42ms/step - loss: 0.0395 Epoch 53/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0394 Epoch 54/100 60/60 [==============================] - 3s 45ms/step - loss: 0.0394 Epoch 55/100 60/60 [==============================] - 3s 48ms/step - loss: 0.0393 Epoch 56/100 60/60 [==============================] - 3s 45ms/step - loss: 0.0393 Epoch 57/100 60/60 [==============================] - 3s 45ms/step - loss: 0.0388 Epoch 58/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0386 Epoch 59/100 60/60 [==============================] - 3s 42ms/step - loss: 0.0386 Epoch 60/100 60/60 [==============================] - 3s 50ms/step - loss: 0.0384 Epoch 61/100 60/60 [==============================] - 3s 45ms/step - loss: 0.0384 Epoch 62/100 60/60 [==============================] - 3s 46ms/step - loss: 0.0382 Epoch 63/100 60/60 [==============================] - 3s 52ms/step - loss: 0.0381 Epoch 64/100 60/60 [==============================] - 3s 50ms/step - loss: 0.0379 Epoch 65/100 60/60 [==============================] - 3s 46ms/step - loss: 0.0379 Epoch 66/100 60/60 [==============================] - 3s 47ms/step - loss: 0.0377 Epoch 67/100 60/60 [==============================] - 3s 49ms/step - loss: 0.0377 Epoch 68/100 60/60 [==============================] - 3s 48ms/step - loss: 0.0376 Epoch 69/100 60/60 [==============================] - 3s 49ms/step - loss: 0.0376 Epoch 70/100 60/60 [==============================] - 3s 53ms/step - loss: 0.0376 Epoch 71/100 60/60 [==============================] - 3s 46ms/step - loss: 0.0375 Epoch 72/100 60/60 [==============================] - 3s 44ms/step - loss: 0.0375 Epoch 73/100 60/60 [==============================] - 3s 48ms/step - loss: 0.0374 Epoch 74/100 60/60 [==============================] - 3s 45ms/step - loss: 0.0372 Epoch 75/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0372 Epoch 76/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0370 Epoch 77/100 60/60 [==============================] - 3s 45ms/step - loss: 0.0370 Epoch 78/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0370 Epoch 79/100 60/60 [==============================] - 3s 42ms/step - loss: 0.0369 Epoch 80/100 60/60 [==============================] - 3s 44ms/step - loss: 0.0368 Epoch 81/100 60/60 [==============================] - 3s 52ms/step - loss: 0.0368 Epoch 82/100 60/60 [==============================] - 3s 49ms/step - loss: 0.0367 Epoch 83/100 60/60 [==============================] - 3s 54ms/step - loss: 0.0366 Epoch 84/100 60/60 [==============================] - 3s 44ms/step - loss: 0.0365 Epoch 85/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0365 Epoch 86/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0365 Epoch 87/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0364 Epoch 88/100 60/60 [==============================] - 3s 42ms/step - loss: 0.0363 Epoch 89/100 60/60 [==============================] - 3s 42ms/step - loss: 0.0364 Epoch 90/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0363 Epoch 91/100 60/60 [==============================] - 3s 44ms/step - loss: 0.0362 Epoch 92/100 60/60 [==============================] - 3s 44ms/step - loss: 0.0361 Epoch 93/100 60/60 [==============================] - 3s 44ms/step - loss: 0.0362 Epoch 94/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0361 Epoch 95/100 60/60 [==============================] - 3s 42ms/step - loss: 0.0361 Epoch 96/100 60/60 [==============================] - 3s 42ms/step - loss: 0.0360 Epoch 97/100 60/60 [==============================] - 3s 42ms/step - loss: 0.0360 Epoch 98/100 60/60 [==============================] - 3s 42ms/step - loss: 0.0360 Epoch 99/100 60/60 [==============================] - 2s 42ms/step - loss: 0.0360 Epoch 100/100 60/60 [==============================] - 3s 43ms/step - loss: 0.0358
同じく、入力(左側)と出力(右側)の結果を確認する。
pred_imgs3 <- Autoencoder3 %>% predict(x_train) pred_imgsR3 <- array_reshape(pred_imgs3, dim=c(dim(pred_imgs3)[1], 28, 28)) dim(pred_imgsR3) par(mfrow=c(3,2)) for (i in 1:6) { m <- sample(1:dim(xtrain)[1], 1, replace = F) display(combine(t(xtrain[m,,]), t(pred_imgsR3[m,,])), method="raster", nx=2, all=TRUE, spacing = 0.01, margin = 2) }
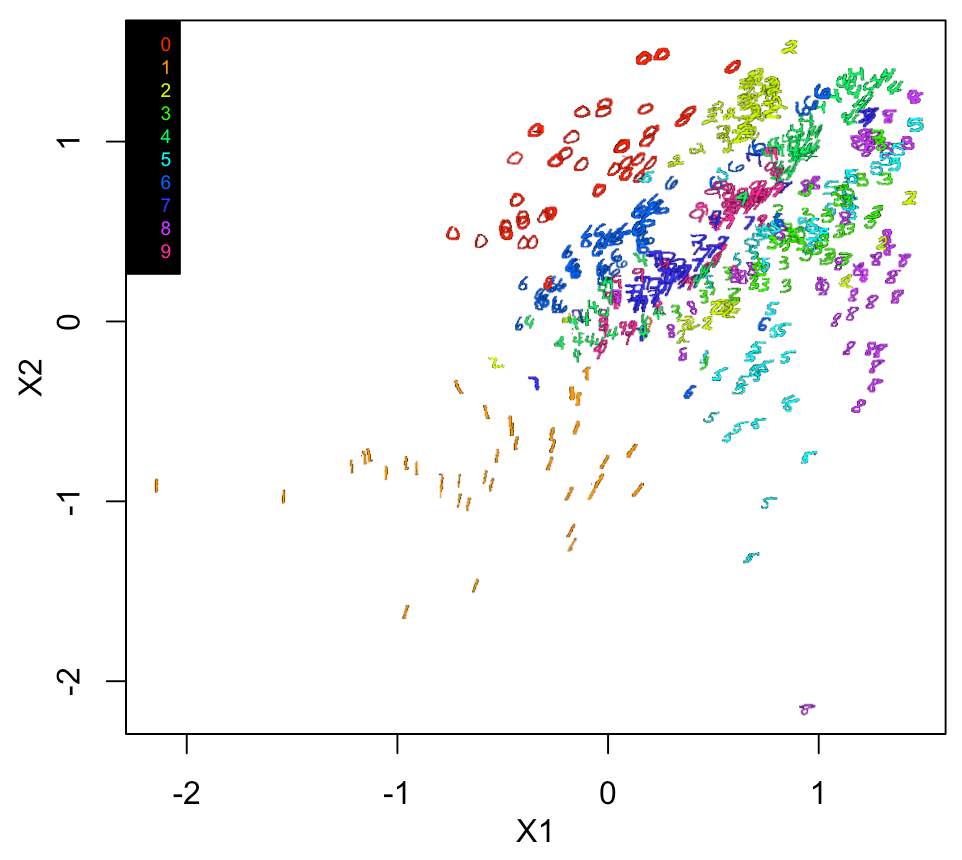
同じく、実際の画像での2Dプロットをやってみると
summary(Autoencoder3) intermediate_layer <- keras_model(inputs = Autoencoder3$input, outputs = get_layer(Autoencoder3, "dense_3")$output) summary(intermediate_layer) intermediate_output <- predict(intermediate_layer, x_train) xy <- data.frame(ytrain, intermediate_output) Sam <- sample(1:nrow(xy), 500, replace = F) xy1 <- xy[Sam,] xy1[,2:3] <- log10(xy1[,2:3]) xy2 <- xtrain[Sam,,] par(mfrow=c(1,1), mai=c(0.75,0.75,0.2,0.2), mgp = c(2,1,0)) plot(xy1[,2:3], pch=21, cex=0.1, col="white") a <- range(xy1[,2][is.finite(xy1[,2])]) b <- range(xy1[,3][is.finite(xy1[,3])]) a1 <- diff(a)*0.015 b1 <- diff(b)*0.015 for(n in 1:nrow(xy1)){ #n <-4 v <- col2rgb(rainbow(10)[xy1[n,1] + 1]) / 255 img = channel(xy2[n,,], 'rgb') img[,,1] <- img[,,1]*v[1] img[,,2] <- img[,,2]*v[2] img[,,3] <- img[,,3]*v[3] ff <- t(as.raster(img)) ff[ff == "#000000"] <- "#00000000" rasterImage(ff, xy1[n,2]-a1, xy1[n,3]-b1, xy1[n,2]+a1, xy1[n,3]+b1) } legend("topleft", legend = c(0:9), cex=0.6, pch=NA, text.col = rainbow(10), bg = "black")
各モデルのlossの比較
| Autoencoder | Final loss |
|---|---|
| Model 1 | 0.0397 |
| Model 2 | 0.0378 |
| Model 3 | 0.0358 |
結果、Science論文を参考にした、3番目のmodelが一番低くなった。
まとめ
いくつかのAutoencoder Modelを、R/Keras/Tensorflowで実装した。
実際、Deep Autoencoderの初期の研究が Scienceに掲載されているとは知らなかった。
いまでも十分使えるし、その派生したアーティテクチャも多数生み出されている。 それだけインパクトがある研究なんだと思う。
R/Kerasを用いたDeep Learningの推薦図書
全コード in Gist
補足
Rstudioにおける、Rセッションのリスタート
.rs.restartR()
Weights全体を取得する場合
autoencoder2_weights <- Autoencoder2 %>% keras::get_weights() str(autoencoder2_weights)
参考文献
https://statslab.eighty20.co.za/posts/autoencoders_keras_r/
tf.keras.utils.plot_model | TensorFlow v2.0.0
Using colorized PNG pictograms in R base plotsryouready.wordpress.com
*1:David E. Rumelhart et al., Learning Internal Representations by Error Propagation, 318-362, 1987., URL: https://apps.dtic.mil/dtic/tr/fulltext/u2/a164453.pdf
*2:Pierre Baldi, Autoencoders, Unsupervised Learning, and Deep Architectures, JMLR: Workshop and Conference Proceedings 27:37–50, 2012., URL: http://proceedings.mlr.press/v27/baldi12a/baldi12a.pdf
*3:G E Hinton, R R Salakhutdinov, Reducing the Dimensionality of Data With Neural Networks, Science 28;313(5786):504-7, 2006., URL: https://science.sciencemag.org/content/313/5786/504
*4:https://www.cs.toronto.edu/~hinton/science.pdf
*5:https://science.sciencemag.org/content/sci/suppl/2006/08/04/313.5786.504.DC1/Hinton.SOM.pdf
*6:https://statslab.eighty20.co.za/posts/autoencoders_keras_r/
*7:https://andrie.github.io/deepviz/index.html
*8:https://bioconductor.org/packages/release/bioc/html/EBImage.html