
- はじめに
- heatmap3パッケージを用いた、ハイカラなヒートマップ表現
- heatmaplyパッケージを使った、ヒートマップ作図
- qtlchartsパッケージのiplotCorrを利用した、相関行列のヒートマップ表現
- iheatmaprパッケージを用いたヒートマップ作成
- corrplotパッケージを用いた、相関ヒートマップ解析
- corrmorantを使った、相関行列マップ
- まとめ
- 参考資料
はじめに
過去に、heatmap.2パッケージと階層的クラスタリングを使って、ヒートマップの描く方を紹介しました。 こんなヒートマップでした。いま思うと、懐かしいような、古めかしい感じがしますね。
今回は、R言語でのヒートマップの特集ということで、 色々なパッケージのヒートマップ表現を調査して、取り上げてみました。
ヒートマップとは、データを可視化するため、 「数値データの高い・低いの表」を「色や濃淡」セルのグリットに変換して表現した視覚化法、あるいはデータ解析法の1つです。 数値の一覧を目視するよりも、データ間の意味や差異がより直感的に導けますし、 「色や濃淡」の曖昧な表現から、データ全体のニュアンスを掴む行為とも言えます。
また、項目間の近縁関係のパターンを強調するために並べられ、しばしばデンドログラムが添えられます。 こうすることで、ヒートマップの見え方が大きく変わり、データからふとした新しい発見にも繋がるかもで、 それは、このヒートマップ解析の興味深いところです。 ヒートマップは、観測値、相関関係、欠損値パターンなどを可視化するために、多くの分野で使用されています。
この記事では、色々なパッケージでのヒートマップの作り方に加えて、 インタラクティブにヒートマップを作成・可視化できるパッケージを紹介します。 全体を通じて、ツール間の比較ができ、良し悪しも感じてもらえるのではと思います。
実行環境
実行環境は以下の通りです。
macOS Big Sur (バージョン11.5.2) MacBook Air (M1, 2020) チップ Apple M1 メモリ 16GB
Rパッケージの読み込み
R環境を立ち上げて、必要なパッケージをインストールしておきます。
#パッケージ・インストール pack <- c("htmlwidgets", "qtlcharts", "qtl", "iheatmapr", "heatmap3", "heatmaply", "remotes", "corrplot") install.packages(pack[!(pack %in% unique(rownames(installed.packages())))]) #ロード for(n in 1:length(pack)){ eval(parse(text = paste0("library(", pack[n], ")"))) }; rm("n", "pack")
heatmap3パッケージを用いた、ハイカラなヒートマップ表現
heatmap2というパッケージがありますが、最近、heatmap3というのも出ているようです。
heatmap3は、改良されたヒートマップパッケージで、Rのオリジナル関数'heatmap'と 完全な互換性で、便利な機能を提供すると説明されています。
それでは、早速、heatmap3のサンプルコードを実行してみます。
heatmap3でのヒートマップ作成(1)シンプルなヒートマップ
この実行例では、乱数を生成して、サンプルデータを作成・利用して、シンプルなヒートマップを作成します。
#乱数でサンプルデータ作成 rnormData <- matrix(rnorm(1000), 40, 25) #head(rnormData) #微調整 rnormData[1:15, seq(6, 25, 2)] <- rnormData[1:15, seq(6, 25, 2)] + 2 rnormData[16:40, seq(7, 25, 2)] = rnormData[16:40, seq(7, 25, 2)] + 4 #列名を与える colnames(rnormData) <- c(paste("Control", 1:5, sep = ""), paste(c("TrtA", "TrtB"), rep(1:10,each=2), sep = "")) #行名を与える rownames(rnormData) <- paste("Probe", 1:40, sep = "") #グループのカラーリング ColSideColors <- cbind(Group1=c(rep("steelblue2",5), rep(c("brown1", "mediumpurple2"),10)), Group2=sample(c("steelblue2","brown1", "mediumpurple2"),25,replace=TRUE)) #特定セルのカラーリング colorCell <- data.frame(row=c(1,3,5),col=c(2,4,6), color=c("green4","black","orange2"),stringsAsFactors=FALSE) #特定セルのハイライト highlightCell <- data.frame(row=c(2,4,6),col=c(1,3,5), color=c("black", "green4","orange2"),lwd=1:3,stringsAsFactors=FALSE) #ヒートマップ実行 heatmap3(rnormData, ColSideColors=ColSideColors, showRowDendro=FALSE, colorCell=colorCell, highlightCell=highlightCell) #注釈 #ColSideColors: 水平サイドバーの色名を含む長さncol(x)の文字ベクトル #showRowDendro: 行のデンドログラムをプロットするかどうかを指定する論理値 #colorCell: 3つの列を持つ data.frame で、どのセルが特定の色で着色されるか #highlightCell: 3列または4列の data.frame で、どのセルが特定の色の矩形でハイライトされるか
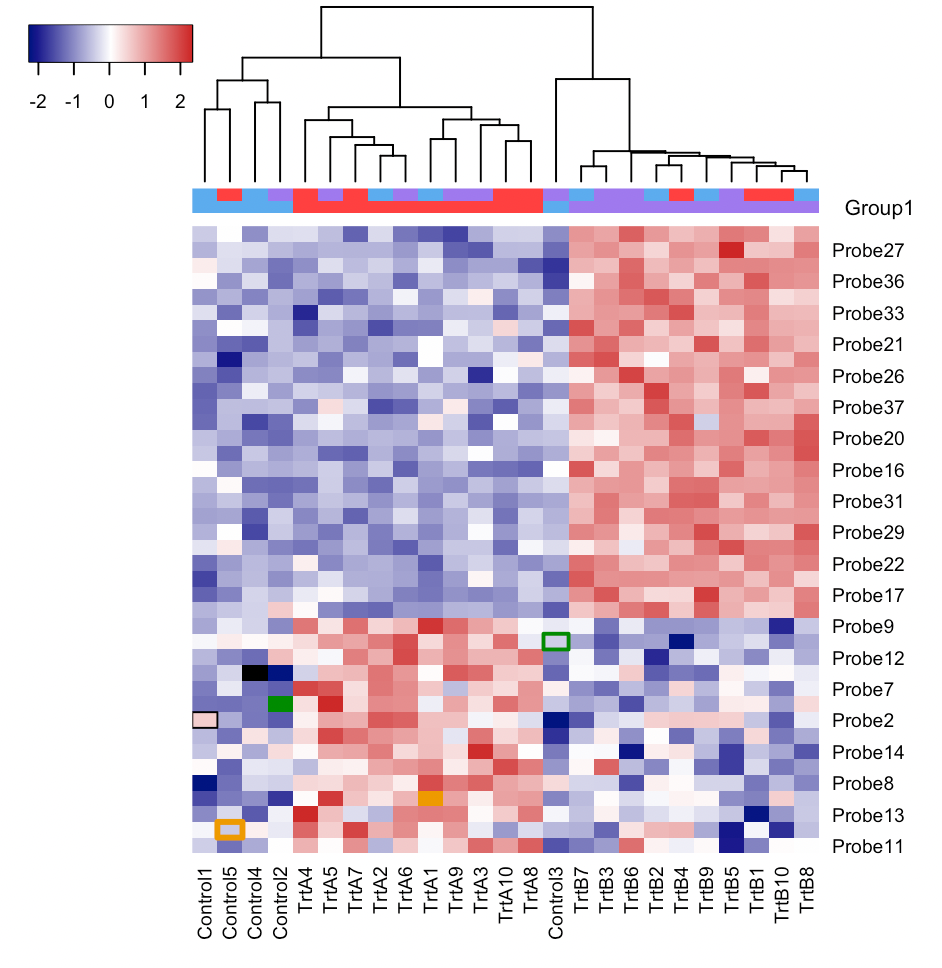
このヒートマップはインタラクティブではない分、見栄えが良いような気がしますね。
heatmap3でのヒートマップ作成(2)少し複雑なヒートマップ
次には、少し複雑なヒートマップの作成を実行します。
#乱数でサンプルデータ作成 ColSideAnn <- data.frame(Information=rnorm(25), Group=c(rep("Control",5),rep(c("TrtA","TrtB"),10)), stringsAsFactors=TRUE) #行名を与える(上記の実行から使用した変数転用) row.names(ColSideAnn) <- colnames(rnormData) #カラーパレット作成 RowSideColors <- colorRampPalette(c("chartreuse4", "white", "firebrick"))(40) result <- heatmap3(rnormData, ColSideCut=1.2, ColSideAnn=ColSideAnn, ColSideFun=function(x) {showAnn(x)}, ColSideWidth=0.8, RowSideColors=RowSideColors, col=colorRampPalette(c("green","black", "red"))(1024), RowAxisColors=1, legendfun=function() {showLegend(legend=c("Low","High"), col=c("chartreuse4","firebrick"))}, verbose=TRUE) #注釈 #ColSideCut: coloumデンドログラムをカットする際に使用される数値 #ColSideAnn: 連続変数と因子変数をアノテーション情報として持つデータフレーム。 #ColSideFun: 列方向に注釈やラベリング図を生成するために使用される関数 #ColSideWidth: 数値列の側面領域の高さ #RowSideColors: (オプション) nrow(x) の長さの文字ベクトル #RowAxisColors: RowSideColors のどの色を行軸のラベルの色として使用するかを示す整数値 #legendfun: 関数で、図の左上に凡例を生成する。
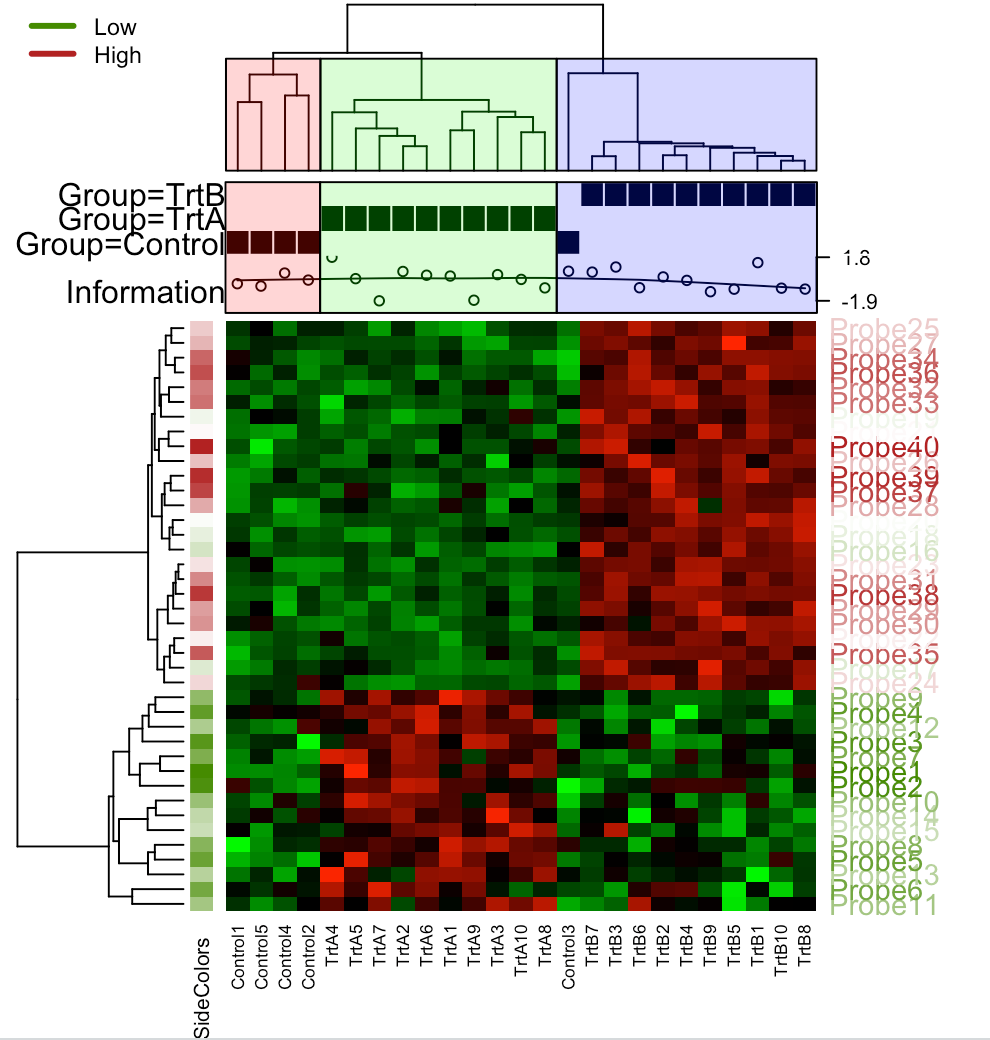
実際、ヒートマップの調整は結構ややこしいです。
heatmaplyパッケージを使った、ヒートマップ作図
次に、heatmaplyパッケージは、ggplot2とplotly.jsのエンジンをベースにして開発されています。 d3heatmapと同様のヒートマップを生成しますが、plotly.jsはより大きなサイズの行列を扱えて、 デンドログラムからのズーム機能の点で優れているようです。
デフォルトのplot_method = "ggplot"の代わりに、
plot_method = "plotly"を選択することもできます。
heatmaplyによる通常のヒートマップ
まずは、mtcarsのデータセットで、heatmaply関数のシンプル実行をしてみます。
#データ表示 head(mtcars) # mpg cyl disp hp drat wt qsec vs am gear carb #Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4 #Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4 #Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1 #Hornet 4 Drive 21.4 6 258 110 3.08 3.215 19.44 1 0 3 1 #Hornet Sportabout 18.7 8 360 175 3.15 3.440 17.02 0 0 3 2 #Valiant 18.1 6 225 105 2.76 3.460 20.22 1 0 3 1 #実行 heatmaply(mtcars)

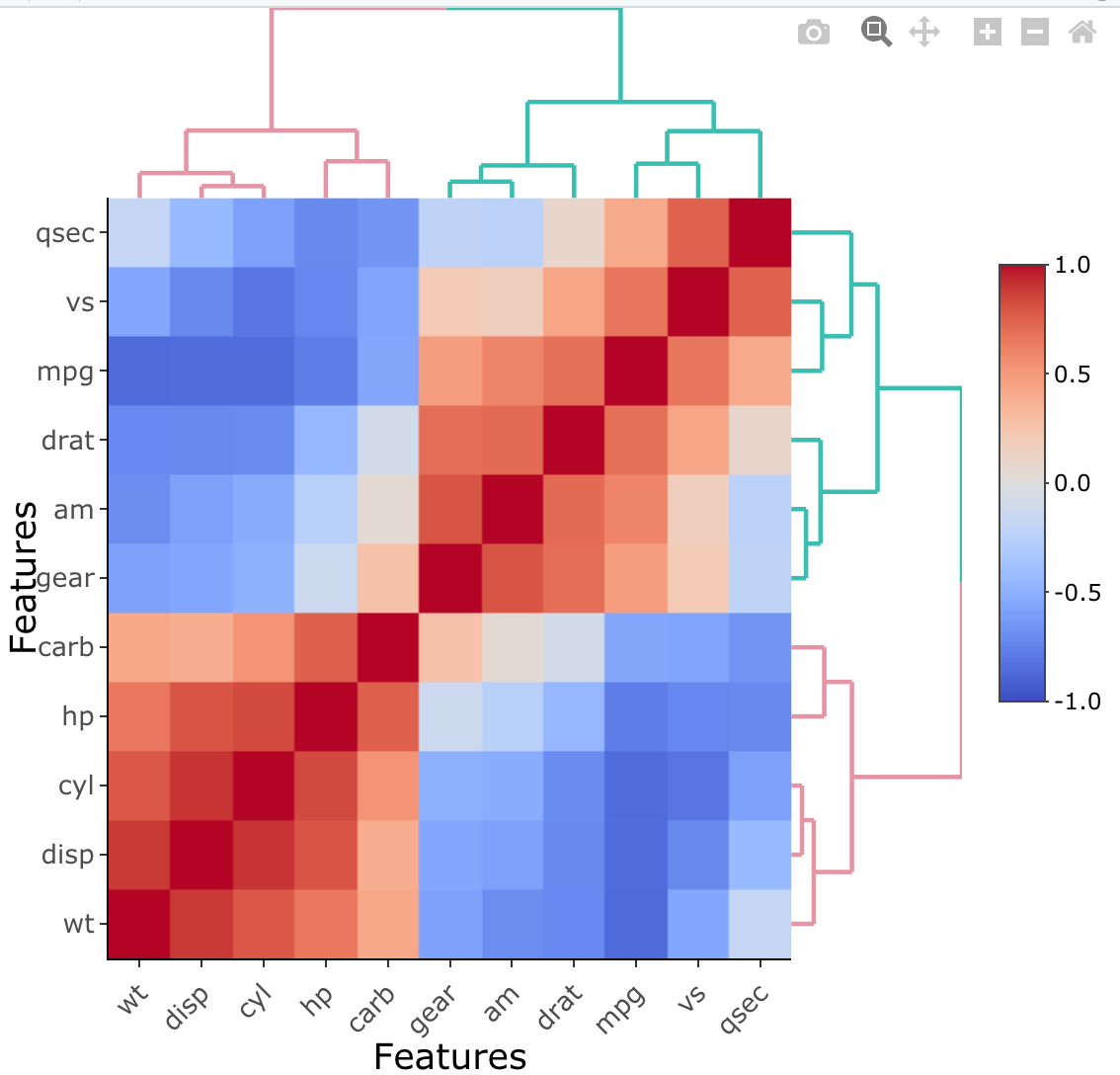
heatmaply_corによる相関ヒートマップ
heatmaply_corは、相関行列に適したヒートマップ関数です。
具体的には、限界値は -1 から 1 に設定され、
カラーパレットはRdBuになっています。
#実行 heatmaply_cor( x = cor(mtcars), xlab = "Features", ylab = "Features", k_col = 2, k_row = 2 )
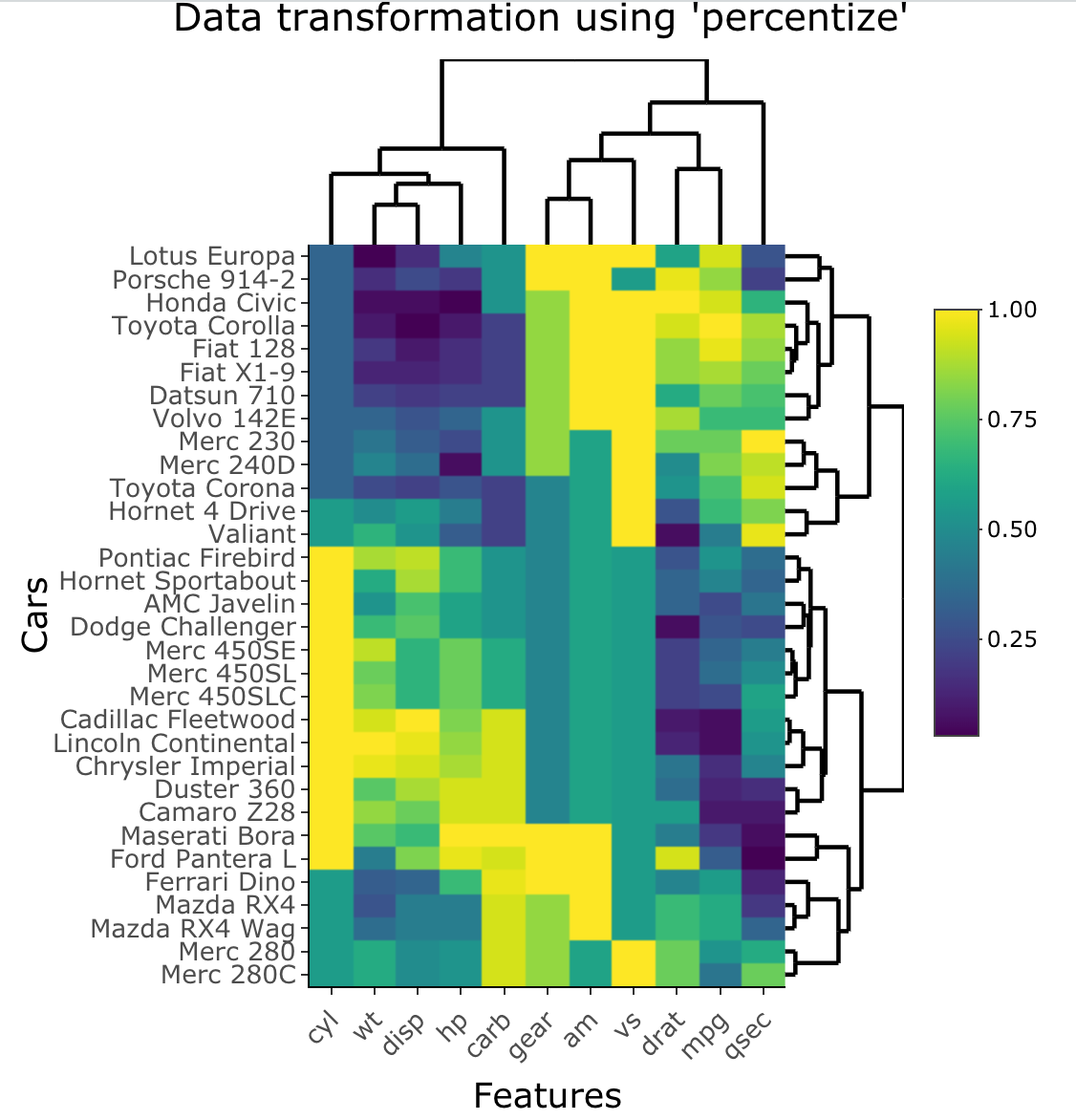
heatmaplyによるpercentizeヒートマップ
percentize関数を使った、経験的パーセンタイル変換は、変数のランクを取ることに似た挙動です。 違いは、変換された変数を比較したり解釈したりするのがよりシンプルになることです。
これは、ヒートマップ(例:heatmaply)において、複数の変数を比較するのに便利な側面があります。
#実行 heatmaply( percentize(mtcars), xlab = "Features", ylab = "Cars", main = "Data transformation using 'percentize'" )
qtlchartsパッケージのiplotCorrを利用した、相関行列のヒートマップ表現
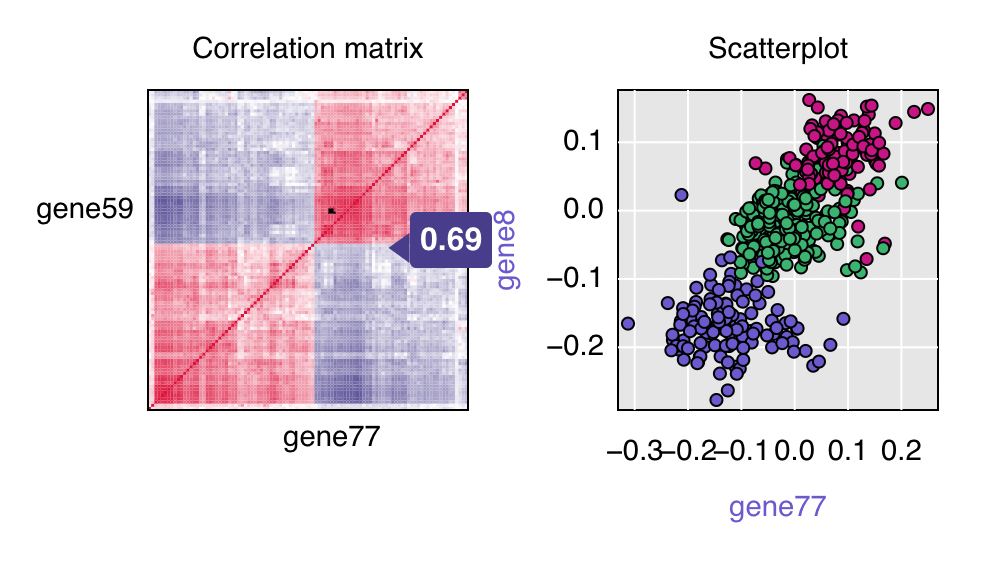
qtlchartsパッケージは、D3.jsを使った、ウェブベースのインタラクティブチャートを提供しています。 パッケージの主な用途は、遺伝子座を実験的に同定するための解析ツールのようです。 その中の1つの関数である、iplotCorrを使用します。
#データ読み込み data(geneExpr) #設定 chartOpts <- list(cortitle="Correlation matrix", scattitle="Scatterplot") #ヒートマップ実行 iplotCorr(mat=geneExpr$expr, group=geneExpr$genotype, reorder=TRUE, chartOpts=chartOpts)
次に、Spearmanの相関行列を使う方法です。
# Spearmanの相関行列計算 corr <- cor(geneExpr$expr, method="spearman", use="pairwise.complete.obs") #設定 chartOpts <- list(cortitle="Spearman matrix", scattitle="Scatterplot") # 階層的クラスタリングによる並び替え o <- hclust(as.dist(1-corr))$order #ヒートマップ実行 iplotCorr(mat=geneExpr$expr[,o], group=geneExpr$genotype, corr=corr[o,o], chartOpts=chartOpts)

iheatmaprパッケージを用いたヒートマップ作成
iheatmaprパッケージは、複雑かつインタラクティブなヒートマップを構築するためのRパッケージです。
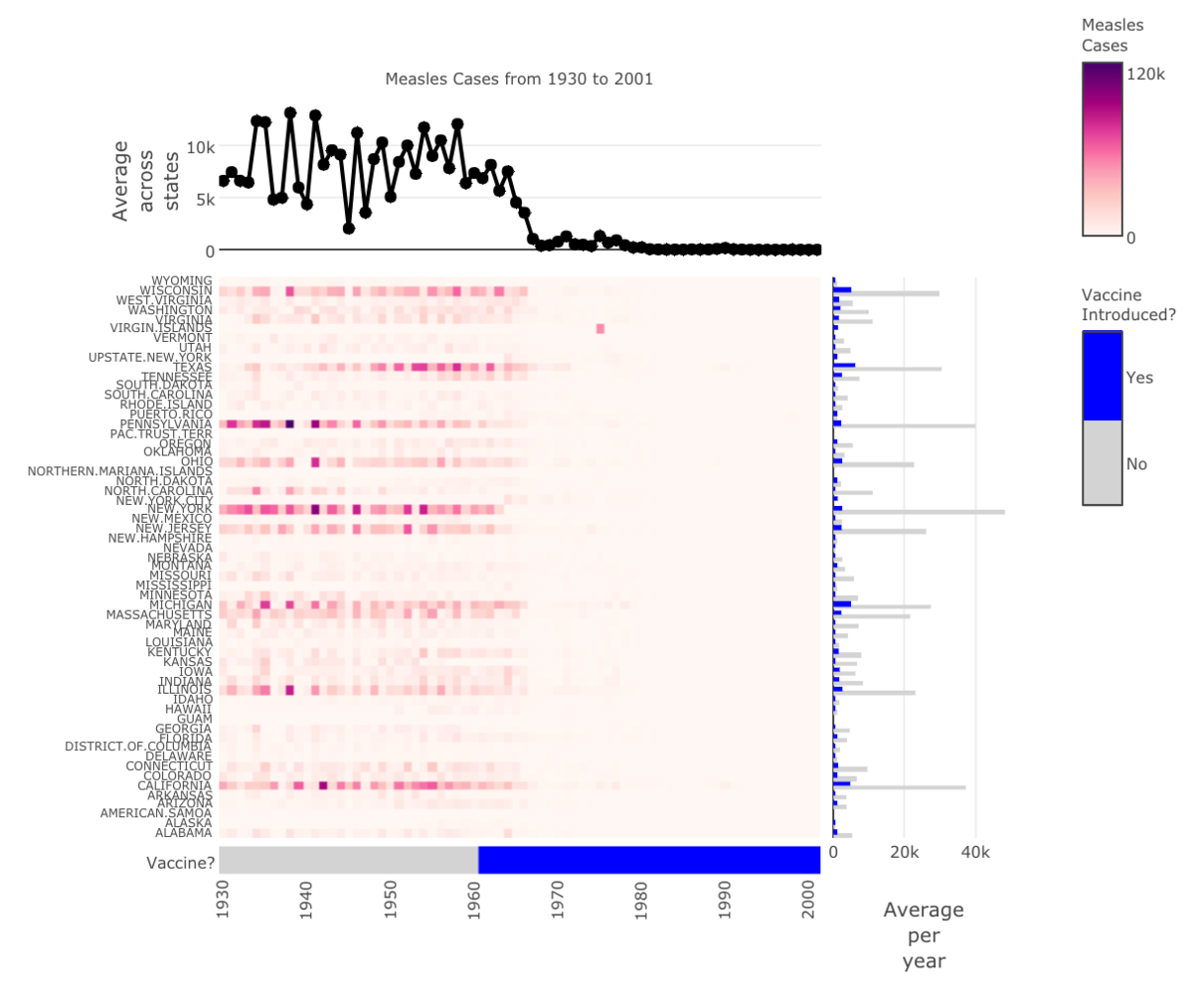
#データ読み込み data(measles, package = "iheatmapr") #ヘッド表示 head(measles) # 1930 1931 1932 1933 1934 #ALABAMA 4389 8934 270 1735 15849 #ALASKA 0 0 0 0 0 #AMERICAN.SAMOA 0 0 0 0 0 #ARIZONA 2107 2135 86 1261 1022 #ARKANSAS 996 849 99 5438 7222 #CALIFORNIA 43585 27807 12618 26551 25650 #実行:メインのみ main_heatmap(measles, name = "Measles<br>Cases", x_categorical = FALSE, layout = list(font = list(size = 8)))
まずは、main_heatmapのみの結果です。

#実行 main_heatmap(measles, name = "Measles<br>Cases", x_categorical = FALSE, layout = list(font = list(size = 8))) %>% add_col_groups(ifelse(1930:2001 < 1961,"No","Yes"), side = "bottom", name = "Vaccine<br>Introduced?", title = "Vaccine?", colors = c("lightgray","blue")) %>% add_col_labels(ticktext = seq(1930,2000,10),font = list(size = 8)) %>% add_row_labels(size = 0.3,font = list(size = 6)) %>% add_col_summary(layout = list(title = "Average<br>across<br>states"), yname = "summary") %>% add_col_title("Measles Cases from 1930 to 2001", side= "top") %>% add_row_summary(groups = TRUE, type = "bar", layout = list(title = "Average<br>per<br>year", font = list(size = 8)))
main_heatmapの結果に、6つのレイアーを付与して、最終的に複雑なヒートマップ図を作成します。
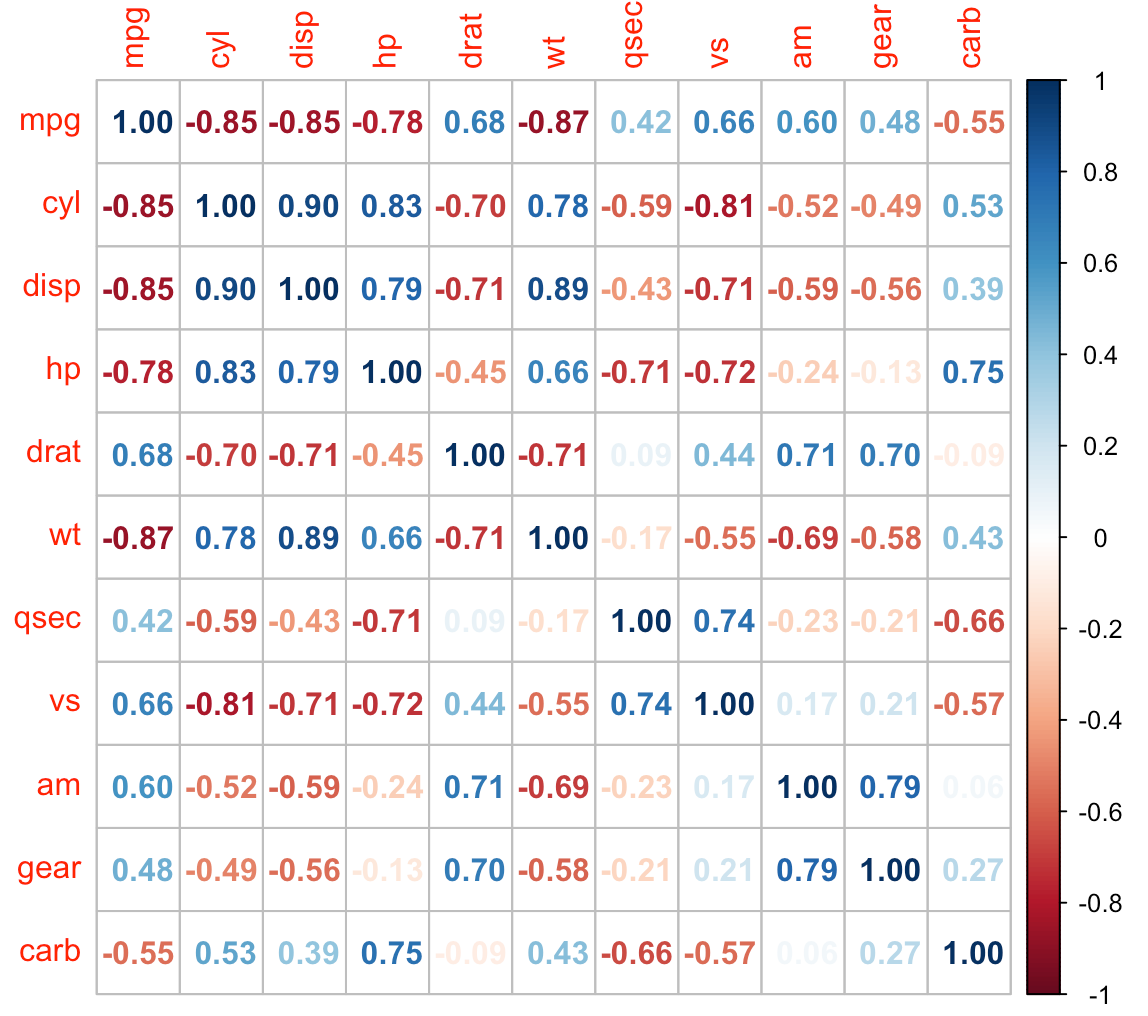
corrplotパッケージを用いた、相関ヒートマップ解析
corrplotは、相関行列を視覚的に探索するツールで、変数間の隠れたパターンを検出するのに役立ちます。また、自動的な変数の並べ替えをサポートしています。また、可視化方法、グラフィックレイアウト、色、凡例、テキストラベルなど、豊富なプロットオプションを提供しています。相関の統計的な有意性を判断するためのp値や信頼区間も提供しています。
#データ準備: mtcarsは自動車の燃費などに関するデータ head(mtcars) # mpg cyl disp hp drat wt qsec vs am gear carb #Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4 #Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4 #Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1 #Hornet 4 Drive 21.4 6 258 110 3.08 3.215 19.44 1 0 3 1 #Hornet Sportabout 18.7 8 360 175 3.15 3.440 17.02 0 0 3 2 #Valiant 18.1 6 225 105 2.76 3.460 20.22 1 0 3 1 #相関の計算 M <- cor(mtcars) #デフォルト相関行列 corrplot(M, method = 'circle')
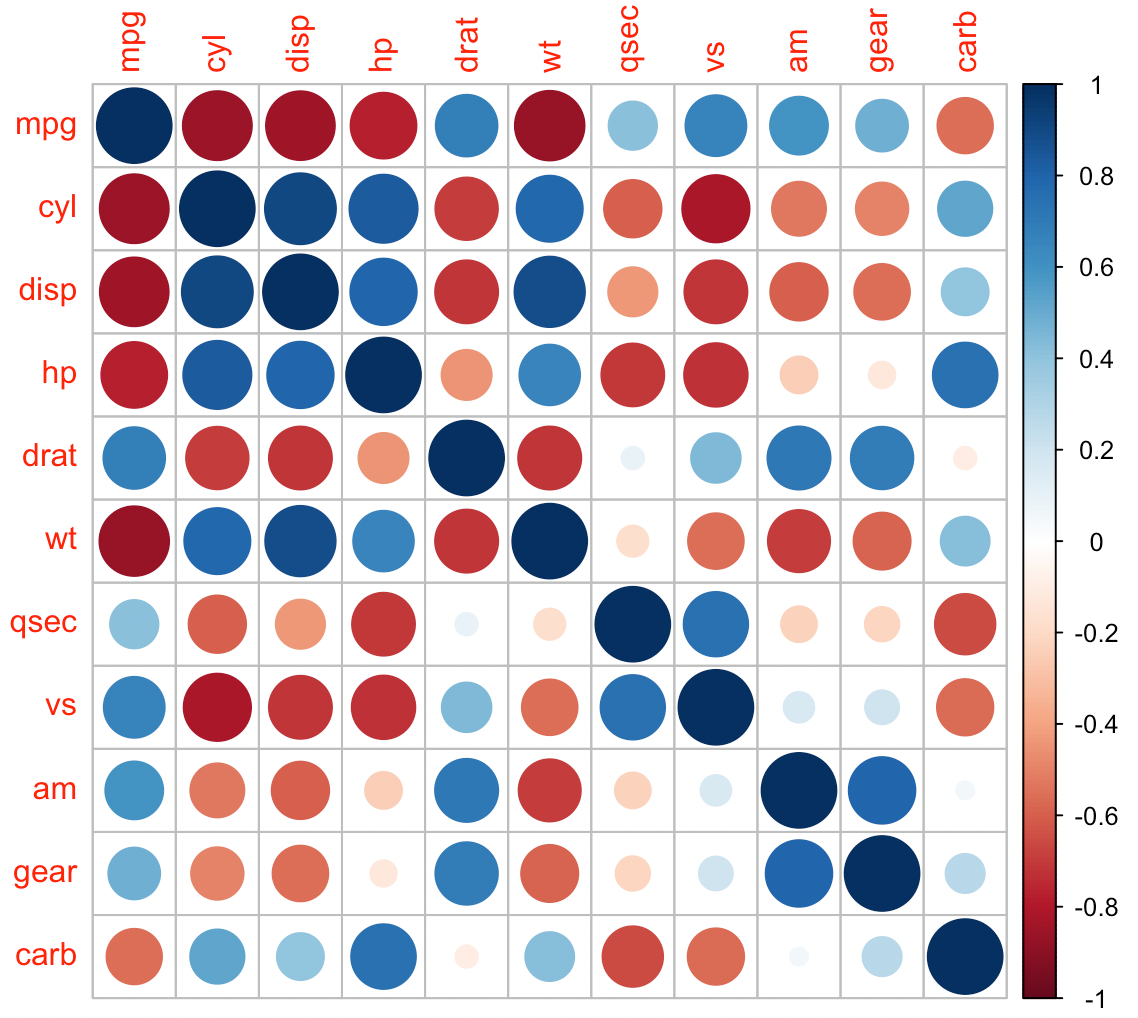
#数字表示有りの相関行列 corrplot(M, method = 'number') # colorful number
corrmorantを使った、相関行列マップ
ヒートマップのパッケージとは言い難いのですが、、このパッケージも追記しておきます。
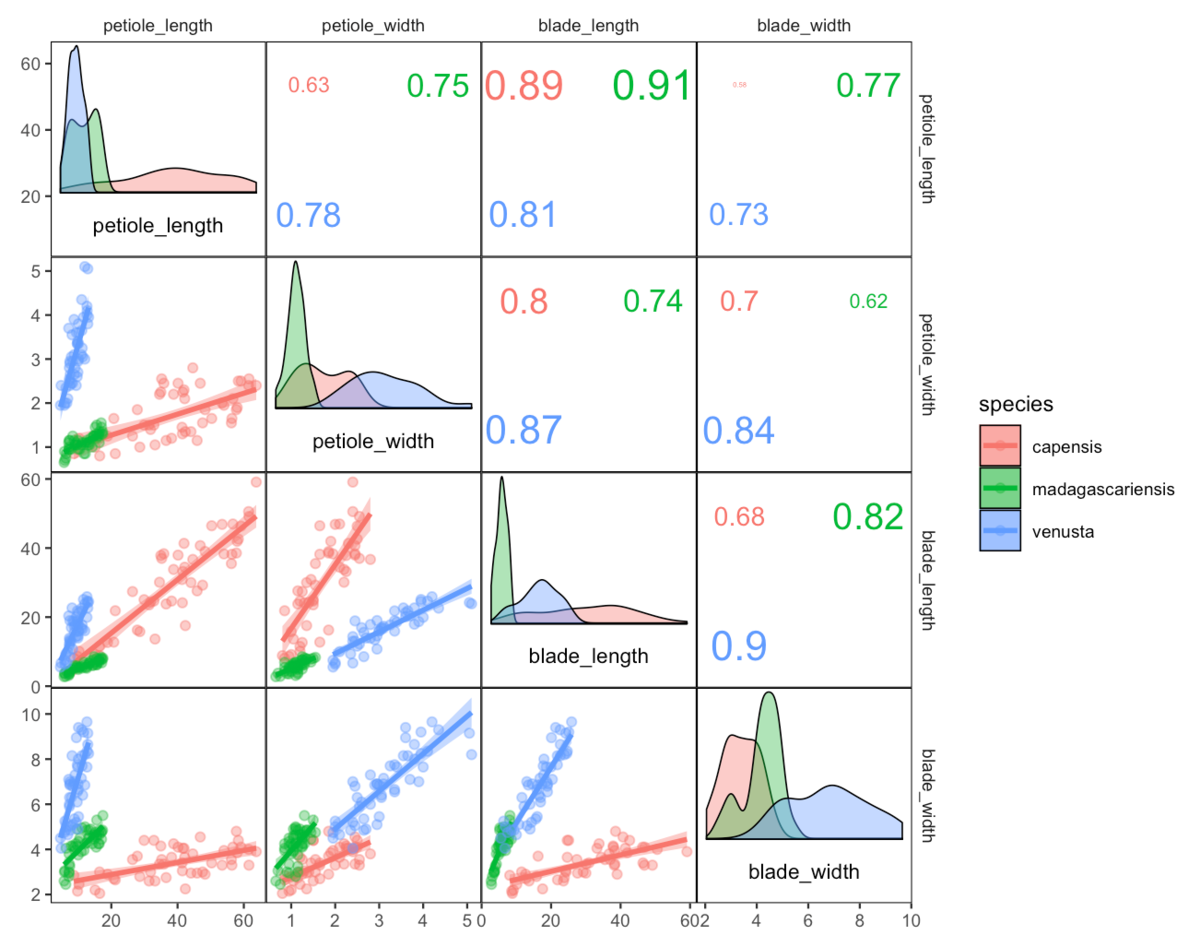
corrmorantパッケージは、通常のggplot2構文で相関行列のプロットを作図できます。 さらに、相関行列に基づく探索的データ解析のための大規模な可視化ツール群を提供しています。
#インストール remotes::install_github("r-link/corrmorant") library(corrmorant) #データ読み込み data(drosera) #表示 head(drosera) # species variety petiole_length petiole_width blade_length blade_width #1 capensis rubra 54.00 1.95 38.30 3.95 #2 capensis rubra 45.90 1.15 27.60 2.95 #3 capensis rubra 21.20 0.85 21.85 2.65 #4 capensis rubra 56.40 1.65 38.20 3.30 #5 capensis rubra 28.60 1.00 15.80 2.80 #6 capensis rubra 32.85 1.50 24.65 4.40 #詳細 str(drosera) #'data.frame': 150 obs. of 6 variables: # $ species : Factor w/ 3 levels "capensis","madagascariensis",..: 1 1 1 1 1 1 1 1 1 1 ... # $ variety : Factor w/ 3 levels "alba","rubra",..: 2 2 2 2 2 2 2 2 2 2 ... # $ petiole_length: num 54 45.9 21.2 56.4 28.6 ... # $ petiole_width : num 1.95 1.15 0.85 1.65 1 1.5 1.85 1.65 2.4 1.5 ... # $ blade_length : num 38.3 27.6 21.9 38.2 15.8 ... # $ blade_width : num 3.95 2.95 2.65 3.3 2.8 4.4 4.8 3.4 3.9 4.4 ...
それでは、droseraのデータを使って、ggcorrmを実行します。 mappingで、colとfillにはspeciesの列を指定します。 2列目のvariety列が無視されるのは、Factorだからですかね。
#実行 ggcorrm(drosera, mapping = aes(col = species, fill = species)) + lotri(geom_smooth(alpha = 0.4, method = "lm")) + lotri(geom_point(alpha = 0.4)) + utri_corrtext(nrow = 2, squeeze = 0.6) + dia_names(y_pos = 0.15, size = 3) + dia_density(lower = 0.3, color = 1, alpha = 0.4) #注釈 #lotri: プロットパネルのサブセットへのggplotレイヤーのマッピングを変更するために使用できるセレクタ関数 #utri_corrtext: プロットの非対角成分での二変量相関の強さをテキストラベルで表示するために使用されます。 #dia_names: プロットの変数名を対角成分の適切な位置にプロットします。 #dia_density: プロットの対角パネルに密度カーブを追加します。
以下のような、結果が得られます。
まとめ
R言語でのヒートマップ特集ということで、最近流行りのヒートマップ表現について調査してみました。 ヒートマップもインタラクティブな可視化が多くなってきている印象ですね。
1つのヒートマップ上に、色々な情報や解析結果を盛り込めるのは良いのですが、 実行コードが複雑で、覚えるのがやや面倒な感じがします。私だけでしょうか。。