- 序章
- ひまわり画像をwikipediaからダウンロードして表示する
- Imageクラスを「行列」に変換する
- RGB成分を3D表示する
- ヒマワリ画像を64x64ピクセルでクロップ(分割)する
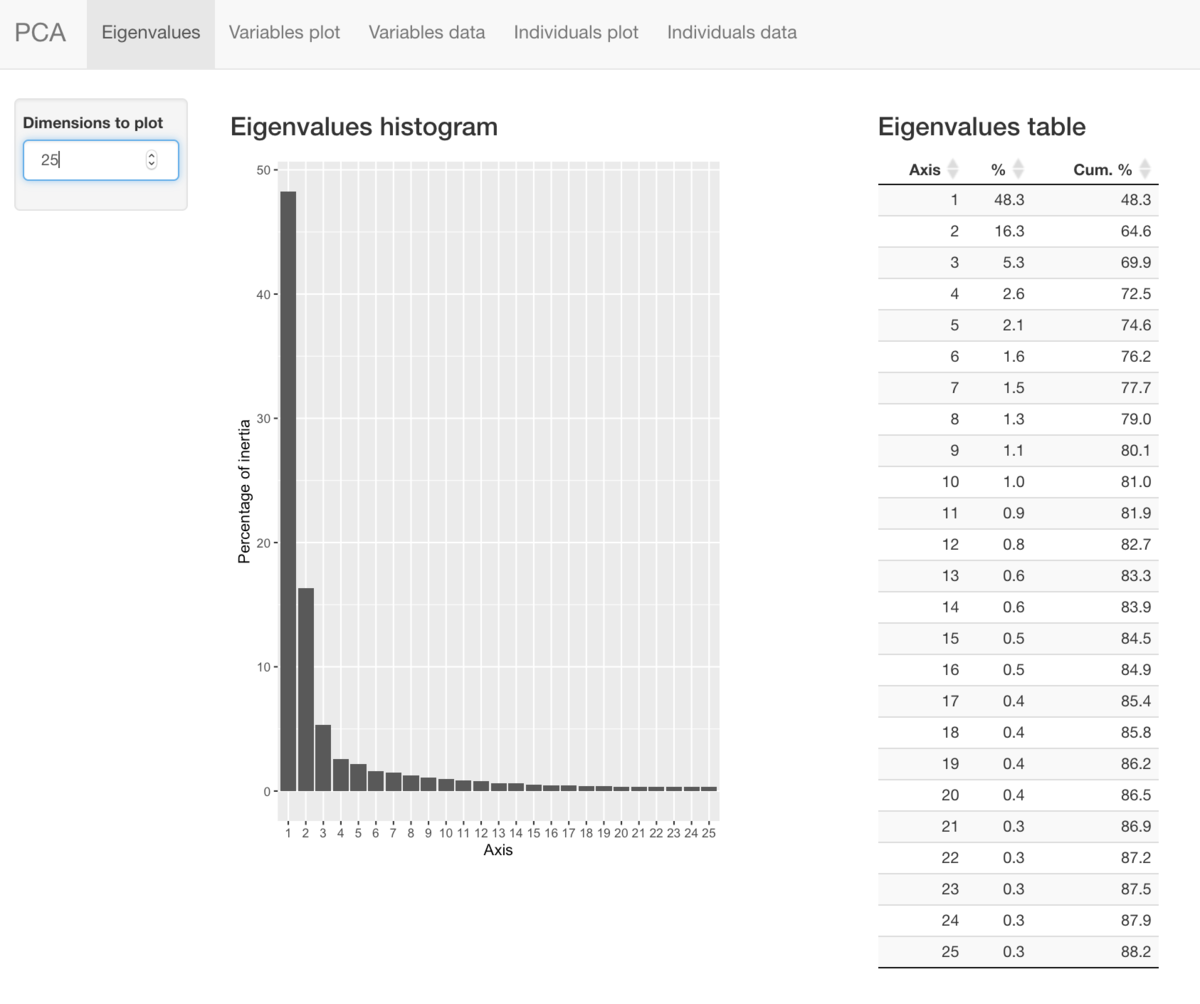
- 主成分分析(PCA)、累積寄与率の計算と可視化
- PCA結果のインタラクティブな可視化
- 主成分得点(Principal component score)の可視化
- 「主成分と変数との相関係数」である因子負荷量(factor loading)の計算と可視化
- 画像データの復元
- まとめ
- 補足
- 参考資料
序章
今回は、Karhunen-Loève変換(KL変換)という手法を使って、 ヒマワリ画像の圧縮を行ってみた*1。
この手法のデフォルトでは、 主成分分析(PCA; Principal Component Analysis)をクロップ画像に適用することで、一度、画像を主成分ベクトルに変換する。 その後、第1~第(p/2)次元の主成分だけを使って、画像を再構成して(PCAの逆計算)、画像の圧縮を行う。
今回使用するヒマワリ画像は、RGB画像である。RGB画像は、R環境で読み込むと、[X, Y, 3]の3次元アレイ(配列)データとして扱われる。Red、Green、Blueがそれぞれ行列データとして扱われ、3次元空間でRGBのピクセル値が表される。
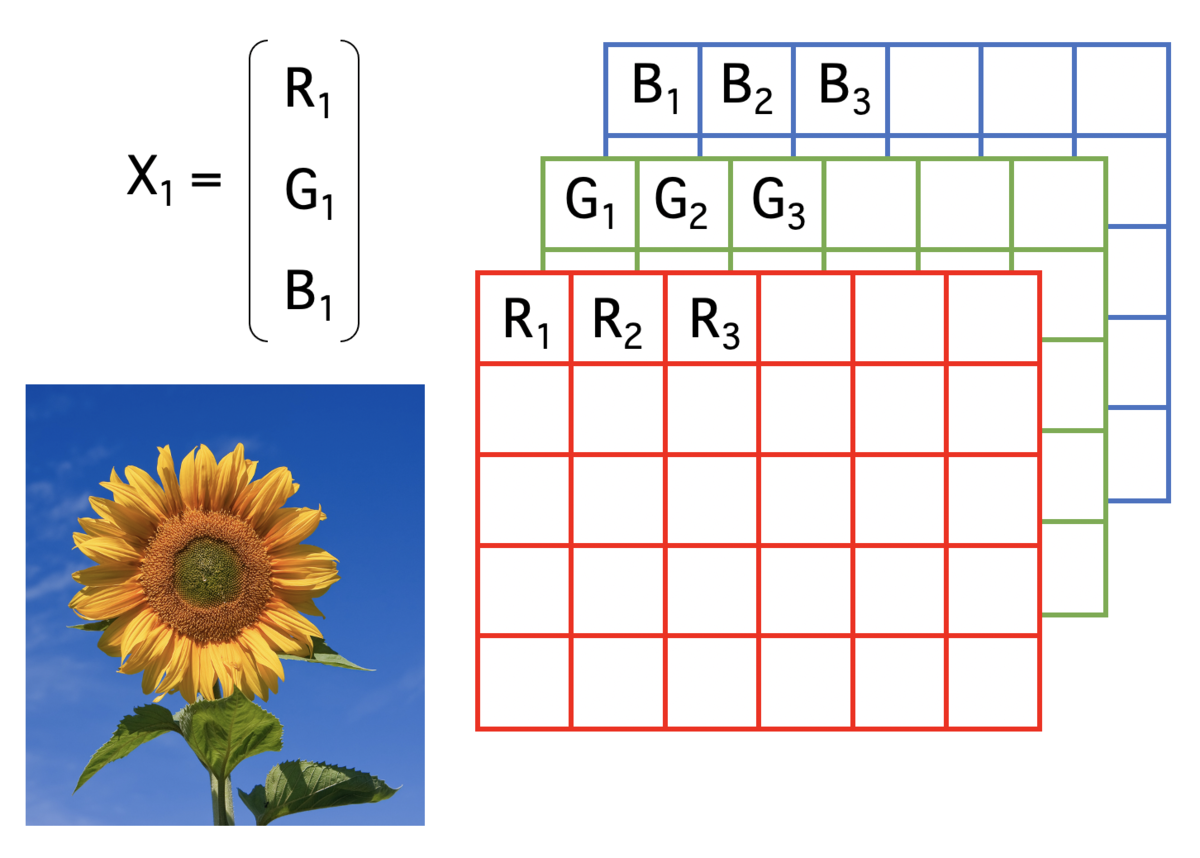
1つの手順として、入力画像をバッチに分割して、 バッチ画像内の画素値をラスタ順に並べて、ベクトル化する。 そのベクトルを積み重ねて、行列のデータセットを作成する。
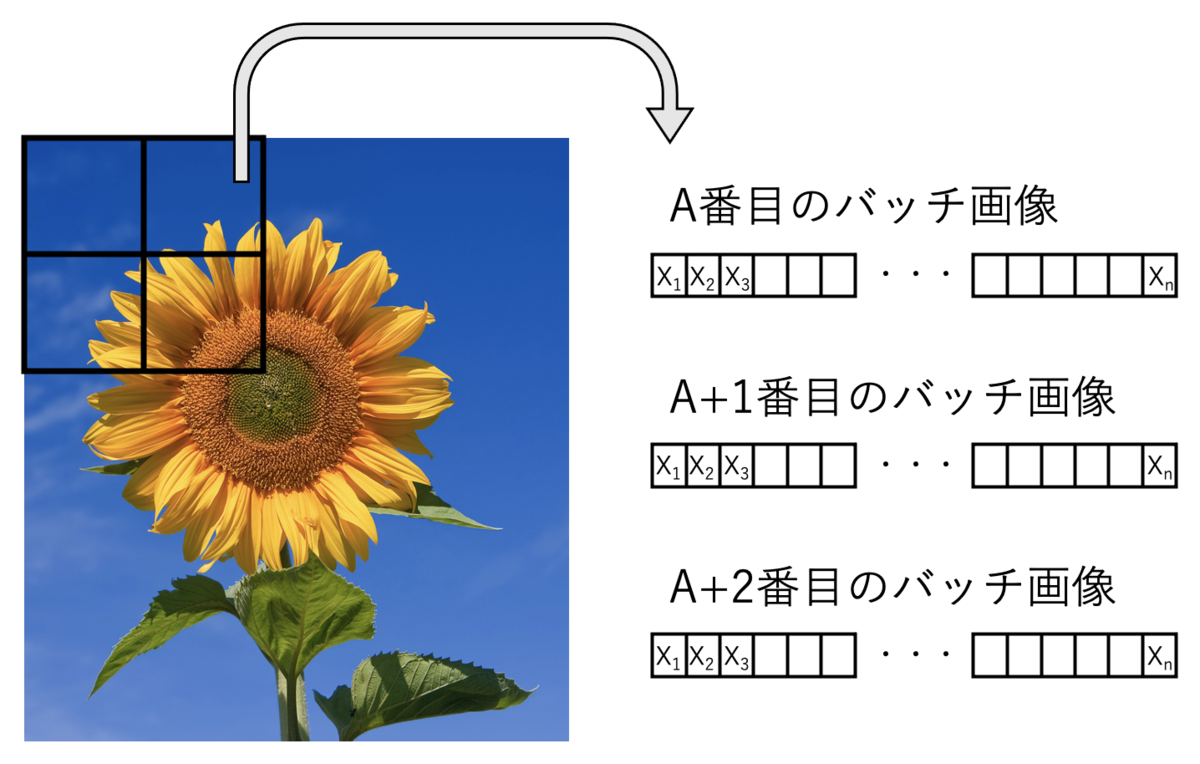
画像をどのくらいのサイズのバッチに分けるか?とか、 何次元までの主成分を用いるか?といったことがポイントになりそうである。
以下に、Rで実際の処理を行ってみた。
関連パッケージのインストール
#EBImageのインストール install.packages("BiocManager") BiocManager::install("EBImage", force = TRUE) #explor & scatterplot3d & animationのインストール install.packages(c("explor", "scatterplot3d", "animation")) #ロード library(EBImage) library(explor) library(scatterplot3d) library(animation)
前回の記事も参照のこと。
ひまわり画像をwikipediaからダウンロードして表示する
download.file関数を使って、Wikipediaからヒマワリの画像をダウンロードする。
そして、EBImageの関数群(readImage & display)を使って、読み込みと表示を行う。
#画像のダウンロード download.file(url = "https://upload.wikimedia.org/wikipedia/commons/4/40/Sunflower_sky_backdrop.jpg", destfile = "sunflower.jpg") #画像の読み込み library(EBImage); options(EBImage.display = "raster") Img <- EBImage::readImage(files="./sunflower.jpg", type = "jpg") #1024x1024にリサイズ Img <- EBImage::resize(Img, w = 1024, h = 1024, filter="bilinear") #画像の表示 EBImage::display(Img) #quartz.save("Img01_sun.png", type="png", dpi=100); dev.off()

Imageクラスを「行列」に変換する
次に、前処理として、 ImageクラスのRGB画像をアレイ(配列)に変換する。 そして、RGBの各成分をベクトル化してから、 RGBの成分を行要素に持つ「行列」に変換する。
#クラス表示 str(Img) #Formal class 'Image' [package "EBImage"] with 2 slots # ..@ .Data : num [1:1024, 1:1024, 1:3] 0.003922 0.006104 0.0069 0.000713 0.002627 ... # ..@ colormode: int 2 # ..$ dim: int [1:3] 1024 1024 3 #アレイに変換 ImgA <- array(Img, dim=dim(Img)) unlist(ImgA)[1:10] # [1] 0.0039215686 0.0061044730 0.0068998412 0.0007129333 0.0026271446 # [6] 0.0039215686 0.0039215686 0.0031252281 0.0000000000 0.0015261519 # 行列に変換 ImgDat <- matrix(unlist(ImgA), ncol = dim(ImgA)[1]*dim(ImgA)[2], byrow = T) ImgDat[,1:10] # [,1] [,2] [,3] [,4] [,5] #[1,] 0.003921569 0.006104473 0.006899841 0.0007129333 0.002627145 #[2,] 0.278431373 0.280614277 0.281409645 0.2752227372 0.277136949 #[3,] 0.654901961 0.657084865 0.657880233 0.6516933254 0.653607537 # [,6] [,7] [,8] [,9] [,10] #[1,] 0.003921569 0.003921569 0.003125228 0.0000000 0.001526152 #[2,] 0.278431373 0.278431373 0.277635032 0.2745098 0.276035956 #[3,] 0.654901961 0.654901961 0.654105620 0.6509804 0.652506544
次に、行列の行要素を戻して、オリジナル画像と各RGB成分を表示させる。
#1枚目: Red成分の分離 ImgDat01 <- Img; ImgDat01[,,c(2:3)] <- 0 #2枚目: Green成分の分離 ImgDat02 <- Img; ImgDat02[,,c(1,3)] <- 0 #3枚目: Blue成分の分離 ImgDat03 <- Img; ImgDat03[,,c(1:2)] <- 0 #RGB成分を分離して表示する EBImage::display(EBImage::combine(Img, ImgDat01, ImgDat02, ImgDat03), nx=2, all=TRUE, spacing = 0.01, margin = 70) #quartz.save("Img02_bunri.png", type="png", dpi=100); dev.off()

RGB成分を3D表示する
#データフレームの列要素に変換する ImgDatRGB <- data.frame(R=as.vector(ImgDat01[,,1]), G=as.vector(ImgDat02[,,2]), B=as.vector(ImgDat03[,,3])) dim(ImgDatRGB) head(ImgDatRGB) #図示 scatterplot3d::scatterplot3d(round(ImgDatRGB, 4), pch=20, color="blue", cex.symbols=0.1, xlab="R", ylab="G", zlab="B",box=F) #quartz.save("Img03_3D.png", type="png", dpi=100); dev.off()
う〜ん、あんまりよく分からない。
ヒマワリ画像を64x64ピクセルでクロップ(分割)する
#パラメータ設定 k <- 64 m <- dim(Img)[1]/k m1 <- c(1:m)*k - k + 1 m2 <- c(1:m)*k #横方向の設定 w1 <- rep(m1, times = m) w2 <- rep(m2, times = m) #縦方向の設定 h1 <- rep(m1, each = length(m1)) h2 <- rep(m2, each = length(m2)) #ピクセス数 p0 <- length(m1)*length(m2) #画像をCropして、ベクトルに変換して重ねていく。 ImgDatCrop <- c() for(n in 1:c(m*m)){ #n <- 2 Crop01 <- unlist(ImgDat01[c(h1[n]:h2[n]),c(w1[n]:w2[n]),1]) Crop02 <- unlist(ImgDat02[c(h1[n]:h2[n]),c(w1[n]:w2[n]),2]) Crop03 <- unlist(ImgDat03[c(h1[n]:h2[n]),c(w1[n]:w2[n]),3]) ImgDatCrop <- rbind(ImgDatCrop, c(Crop01, Crop02, Crop03)) } #詳細 dim(ImgDatCrop) #[1] 256 12288 ImgDatCrop[1:3,1:10] # [,1] [,2] [,3] [,4] [,5] #[1,] 0.003921569 0.006104473 0.006899841 0.0007129333 0.002627145 #[2,] 0.023931956 0.026674185 0.027450980 0.0235294118 0.022355663 #[3,] 0.003193934 0.000000000 0.000000000 0.0000000000 0.000000000 # [,6] [,7] [,8] [,9] [,10] #[1,] 0.003921569 0.003921569 0.003125228 0.00000000 0.001526152 #[2,] 0.021938189 0.028931143 0.044128429 0.05367229 0.058823529 #[3,] 0.001536227 0.003921569 0.002798294 0.00000000 0.000000000 #クロップ画像の表示 par(mfcol = c(length(m1), length(m2)), mai = c(0.1, 0.1, 0.1, 0.1)) for(n in 1:p0){ a <- EBImage::Image(array(ImgDatCrop[n,], dim=c(k, k, 3)), colormode = "Color") EBImage::display(EBImage::rotate(a, angle=0)) } #quartz.save("Img04.png", type="png", dpi=100); dev.off() #saveRDS(ImgDatCrop, "ImgDatCrop.Rds")

このステップで、ベクトル画像を元の64x64画像に戻して、タイル状に表示した。
主成分分析(PCA)、累積寄与率の計算と可視化
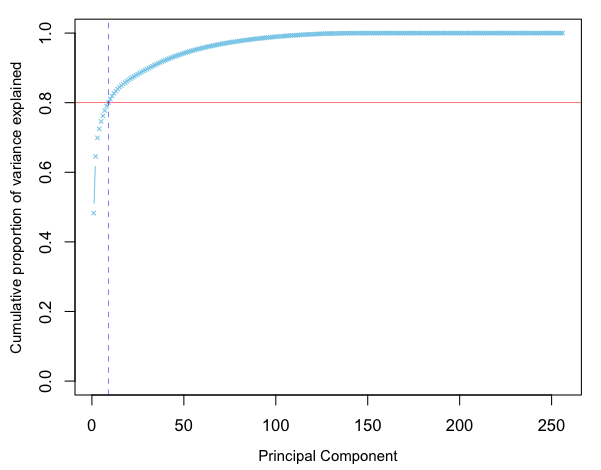
続いて、クロップ画像の相関行列に対して、 PCAを実行して、累積寄与率などの計算や可視化を行う。
#再読み込みの場合 #ImgDatCrop <- readRDS("ImgDatCrop.Rds") #次元数 dim(ImgDatCrop) #Q-mode PCA: 相関行列 pca <- prcomp(ImgDatCrop, scale = TRUE) #固有値(Standard deviation)、寄与率(Proportion of Variance)、累積寄与率(Cumulative Proportion)の表示 round(summary(pca)$importance, 2)[,1:10] # PC1 PC2 PC3 PC4 PC5 PC6 PC7 PC8 PC9 PC10 #Standard deviation 77.00 44.76 25.62 17.82 16.24 14.03 13.58 12.42 11.47 11.01 #Proportion of Variance 0.48 0.16 0.05 0.03 0.02 0.02 0.01 0.01 0.01 0.01 #Cumulative Proportion 0.48 0.65 0.70 0.72 0.75 0.76 0.78 0.79 0.80 0.81 #主成分分析の主成分(固有ベクトル)の表示(一部) round(pca$rotation, 3)[c(1:5),c(1:10)] # PC1 PC2 PC3 PC4 PC5 PC6 PC7 PC8 PC9 PC10 #[1,] 0.01 0.000 -0.012 0.008 -0.008 -0.006 -0.009 -0.011 0.003 -0.02 #[2,] 0.01 0.001 -0.012 0.009 -0.008 -0.007 -0.007 -0.009 0.000 -0.02 #[3,] 0.01 0.001 -0.012 0.009 -0.009 -0.008 -0.005 -0.008 0.001 -0.02 #[4,] 0.01 0.001 -0.012 0.008 -0.010 -0.008 -0.004 -0.009 0.002 -0.02 #[5,] 0.01 0.001 -0.012 0.009 -0.010 -0.006 -0.004 -0.011 0.002 -0.02 #主成分得点(一部) round(pca$x, 3)[c(1:5),c(1:10)] # PC1 PC2 PC3 PC4 PC5 PC6 PC7 PC8 PC9 PC10 #[1,] -85.099 -41.512 6.937 -0.125 0.823 -0.755 0.065 0.894 -1.743 1.595 #[2,] -84.157 -37.342 -1.268 -0.003 3.570 -1.325 1.064 1.971 0.358 1.698 #[3,] -85.098 -33.581 3.612 0.768 2.403 -1.103 2.272 2.372 -2.263 1.970 #[4,] -70.808 -9.993 19.623 -1.219 0.390 -4.685 -1.409 1.029 3.036 0.987 #[5,] -67.116 -7.996 -0.804 -1.582 0.146 0.598 -2.740 -0.015 1.285 0.306 #Cumulative Proportion (累積寄与率)の計算 vars <- apply(pca$x, 2, var) result <- cumsum(vars / sum(vars)) par(mgp=c(2.5, 1, 0), mai = c(0.75, 0.75, 0.2, 0.2)) plot(result, type="b", pch=4, col="skyblue", xlab="Principal Component", ylim=c(0,1), ylab="Cumulative proportion of variance explained", cex=0.5, cex.lab=0.9) abline(h=0.8, col="red", lwd=0.5); abline(v=sum(result < 0.8)+1, col="blue", lty=2, lwd=0.5) #quartz.save("Img05_CumulativeProportion.png", type="png", dpi=100); dev.off()
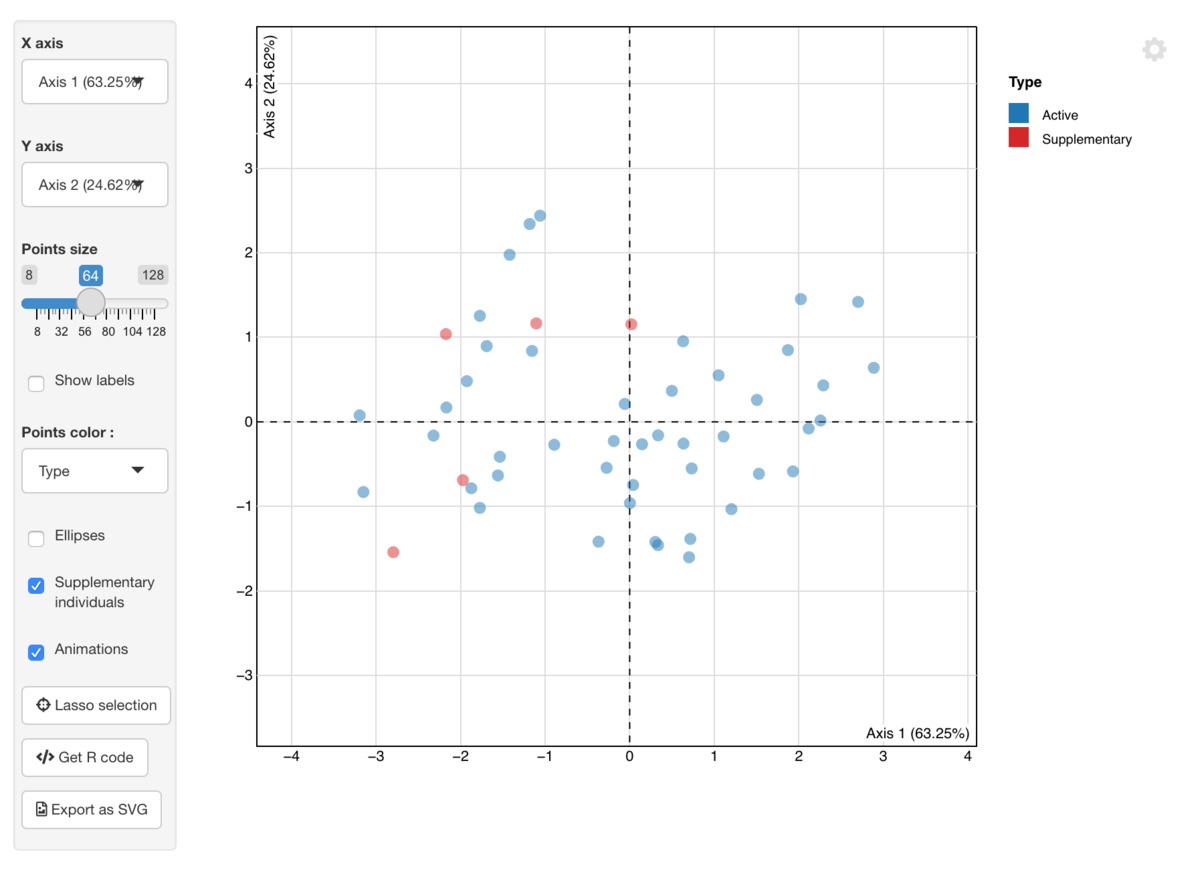
PCA結果のインタラクティブな可視化
explorパッケージは、解析結果探索のためのインターフェース を提供します。
explorを実行するとShinyアプリを起動して、 PCAの解析結果をインタラクティブに可視化・探索できます。
library(explor) #インタラクティブ表示 explor::explor(pca)
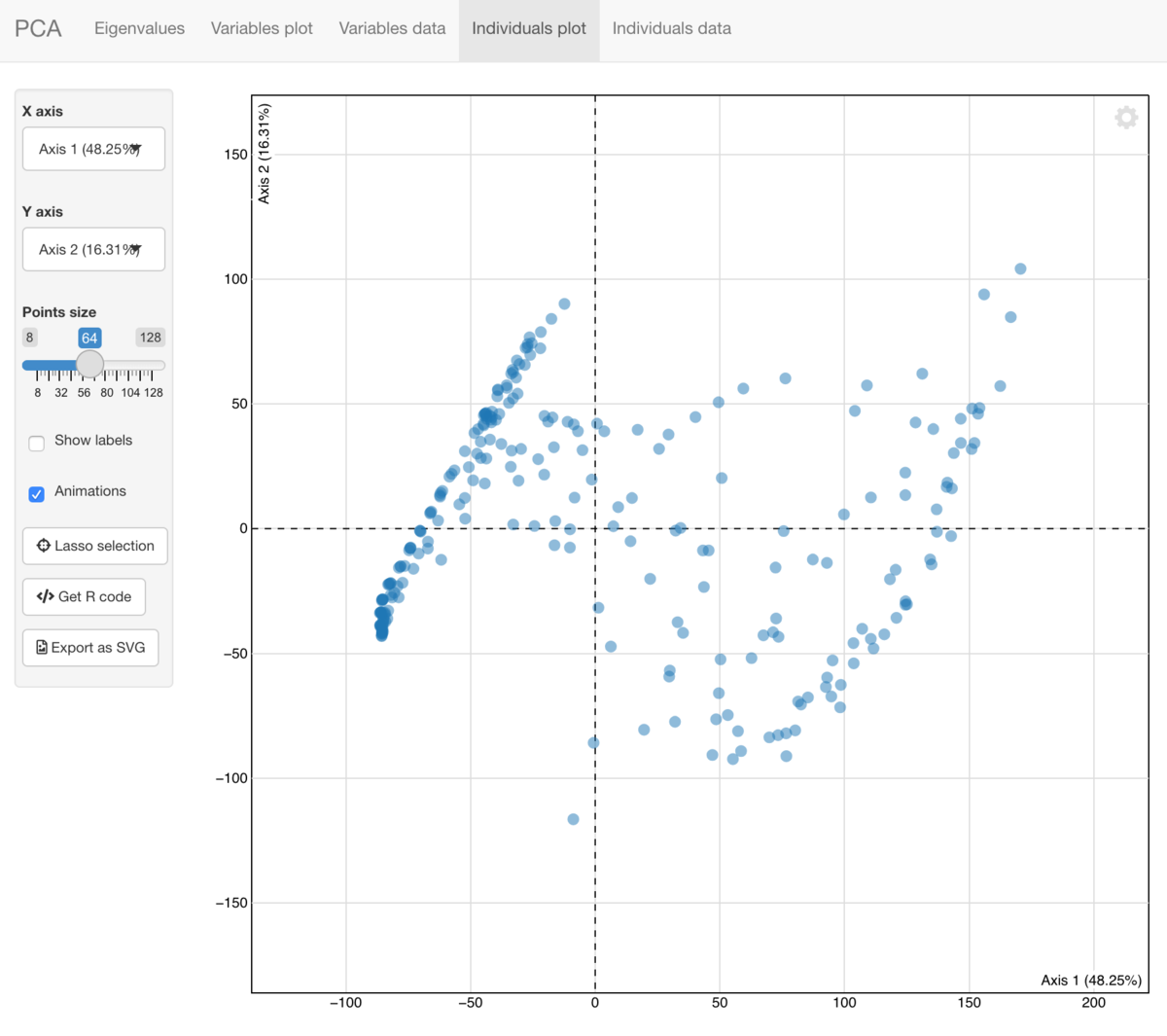
shinyを使うけど、これの可視化ツールは、結構便利かも。
主成分得点(Principal component score)の可視化
次に、主成分得点を二次元にマッッピングする。
#ロード library(EBImage); options(EBImage.display = "raster") #主成分得点: 元の情報から情報損失量を除いた、新しい情報量を意味する pcaScore <- round(pca$x, 4) str(pcaScore) # num [1:256, 1:256] -85.1 -84.2 -85.1 -70.8 -67.1 ... # - attr(*, "dimnames")=List of 2 # ..$ : NULL # ..$ : chr [1:256] "PC1" "PC2" "PC3" "PC4" ... #最小値を0、最大値を1とする正規化 pcaScore0 <- scale(pcaScore, center = apply(pcaScore, 2, min), scale = (apply(pcaScore, 2, max) - apply(pcaScore, 2, min))) #displayでの作図 #b <- EBImage::Image(array(pcaScore0, dim=c(p0, p0, 1)), colormode = "Grayscale") #EBImage::display(EBImage::rotate(b, angle=0)) #imageでの作図 par(mgp=c(1, 1, 0), cex=1.25) graphics::image(x=1:p0, y=1:p0, z=matrix(rev(unlist(pcaScore0[,-p0])), nrow = 256, byrow = F), xaxt = "n", yaxt = "n", xlab="PC (1-256)", ylab="Components") #quartz.save("Img06_pcaScore0.png", type="png", dpi=100); dev.off()
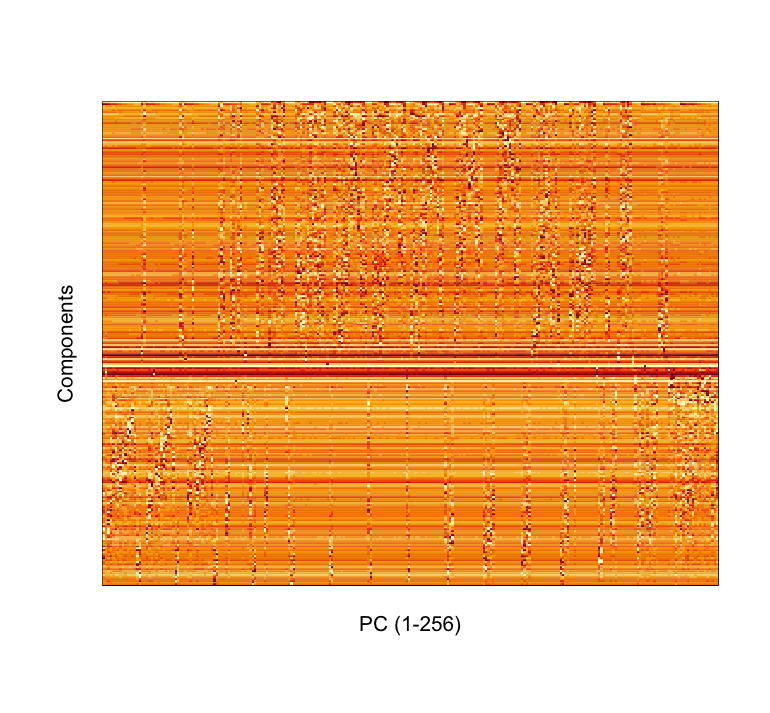
やっぱ、列ベクトルのヒートマップだけ見ても、よくわからないね。
そこで、PC1成分を棒グラフにしてみる。
head(pcaScore0)[,1:5] # PC1 PC2 PC3 PC4 PC5 #[1,] 0.005025779 0.3398974 0.4935447 0.4058873 0.5197696 #[2,] 0.008690831 0.3587962 0.4509923 0.4067744 0.5419167 #[3,] 0.005027335 0.3758440 0.4763016 0.4124084 0.5325050 #[4,] 0.060633086 0.4827584 0.5593437 0.3978971 0.5162717 #[5,] 0.074997179 0.4918104 0.4533958 0.3952488 0.5143074 #[6,] 0.075153602 0.5043891 0.4422169 0.3571957 0.5133769 #PC1の棒グラフ barplot(pcaScore0[,1]) #quartz.save("Img07_pcaScore0.png", type="png", dpi=100); dev.off()
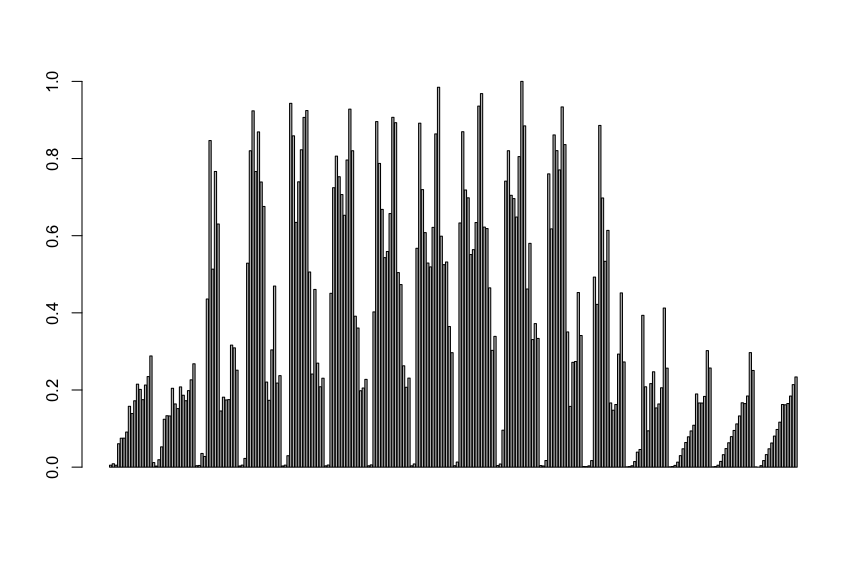
こっちの方がまだ分かりやすい。 ただ、何の成分が影響するかはイマイチ分からない。
「主成分と変数との相関係数」である因子負荷量(factor loading)の計算と可視化
#EBImageのロード library(EBImage); options(EBImage.display = "raster") #因子負荷量(factor loading) or 主成分負荷量(principal component loading): #計算方法: 固有ベクトル(result$rotation)と対応した固有値の平方根(result$sdev)との積をとる pca01 <- sweep(x=pca$rotation, MARGIN=2, STATS=pca$sdev, FUN="*") #第25主成分までの結果を可視化する par(mfrow = c(5, 5), mai = c(0.1, 0.05, 0.1, 0.05)) for(N in 1:25){ b <- EBImage::Image(array(pca01[,N], dim=c(k, k, 3)), colormode = "Color") EBImage::display(b) text(x = 5, y = 10, label = paste0("PC", N), adj = c(0,0), col = "white", cex = 1) } #quartz.save("Img08.png", type="png", dpi=100); dev.off()
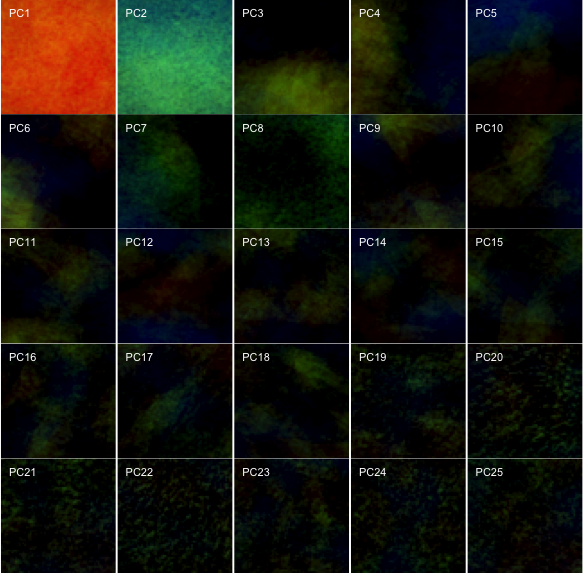
主成分負荷量まで計算して、二次元にマップする。 画像の特徴が何だかそれっぽく見えてくる。
画像データの復元
まずは、全データでのPCAの逆計算を行う。
#相関行列PCAの逆計算 pca00 <- t(t(pca$x %*% t(pca$rotation)) * pca$scale + pca$center) #元データ head(round(ImgDatCrop, 4))[c(1:5),c(1:5)] # [,1] [,2] [,3] [,4] [,5] #[1,] 0.0039 0.0061 0.0069 0.0007 0.0026 #[2,] 0.0239 0.0267 0.0275 0.0235 0.0224 #[3,] 0.0032 0.0000 0.0000 0.0000 0.0000 #[4,] 0.0575 0.0531 0.0504 0.0474 0.0370 #[5,] 0.1019 0.1019 0.1020 0.1020 0.1020 #逆計算の結果 head(round(pca00, 4))[c(1:5),c(1:5)] # [,1] [,2] [,3] [,4] [,5] #[1,] 0.0039 0.0061 0.0069 0.0007 0.0026 #[2,] 0.0239 0.0267 0.0275 0.0235 0.0224 #[3,] 0.0032 0.0000 0.0000 0.0000 0.0000 #[4,] 0.0575 0.0531 0.0504 0.0474 0.0370 #[5,] 0.1019 0.1019 0.1020 0.1020 0.1020 #一致数 table(round(ImgDatCrop, 4) == round(pca00, 4)) # FALSE TRUE # 3 3145725
元データと逆計算の結果は、少し誤差が出るみたいだけど、ほぼほぼ一致した。
ここで、%*%の演算子は、行列の積を求める。
続いて、第1主成分のみ、第1〜5主成分、第1〜10主成分、第1〜25主成分を使って 画像の圧縮を行い、復元画像の結果を可視化する。
第1主成分のみ
主成分スコア pca$xの対象列以外(はじめは、第1主成分以外)をゼロにして、PCAの逆計算を行う。
#第1主成分のみ pcaP <- pca pcaP$x[,-c(1)] <- 0 #相関行列PCAの逆計算 pca01 <- t(t(pcaP$x %*% t(pcaP$rotation)) * pcaP$scale + pcaP$center) #クロップ画像の表示 par(mfcol = c(length(m1), length(m2)), mai = c(0.1, 0.1, 0.1, 0.1)) for(n in 1:p0){ a <- EBImage::Image(array(pca01[n,], dim=c(k, k, 3)), colormode = "Color") EBImage::display(a) } #quartz.save("Img09_PCA.png", type="png", dpi=100); dev.off()

第1-5主成分
#第1-5主成分 pcaP <- pca pcaP$x[,-c(1:5)] <- 0 #相関行列PCAの逆計算 pca01 <- t(t(pcaP$x %*% t(pcaP$rotation)) * pcaP$scale + pcaP$center) #クロップ画像の表示 par(mfcol = c(length(m1), length(m2)), mai = c(0.1, 0.1, 0.1, 0.1)) for(n in 1:p0){ a <- EBImage::Image(array(pca01[n,], dim=c(k, k, 3)), colormode = "Color") EBImage::display(a) } #quartz.save("Img10_PCA.png", type="png", dpi=100); dev.off()

第1-10主成分
#第1-10主成分 pcaP <- pca pcaP$x[,-c(1:10)] <- 0 #相関行列PCAの逆計算 pca01 <- t(t(pcaP$x %*% t(pcaP$rotation)) * pcaP$scale + pcaP$center) #クロップ画像の表示 par(mfcol = c(length(m1), length(m2)), mai = c(0.1, 0.1, 0.1, 0.1)) for(n in 1:p0){ a <- EBImage::Image(array(pca01[n,], dim=c(k, k, 3)), colormode = "Color") EBImage::display(a) } #quartz.save("Img11_PCA.png", type="png", dpi=100); dev.off()

第1-25主成分
#第1-25主成分 pcaP <- pca pcaP$x[,-c(1:25)] <- 0 #相関行列PCAの逆計算 pca01 <- t(t(pcaP$x %*% t(pcaP$rotation)) * pcaP$scale + pcaP$center) #クロップ画像の表示 par(mfcol = c(length(m1), length(m2)), mai = c(0.1, 0.1, 0.1, 0.1)) for(n in 1:p0){ a <- EBImage::Image(array(pca01[n,], dim=c(k, k, 3)), colormode = "Color") EBImage::display(a) } #quartz.save("Img12_PCA.png", type="png", dpi=100); dev.off()

次元圧縮のアニメーション
最後に、主成分数を変えて、計算結果の画像をアニメーションにしてみた。
library(EBImage); options(EBImage.display = "raster") install.packages("animation"); library(animation) #主成分数 Num <- 100 #アニメーション作成 animation::saveGIF({ for(m in 1:Num){ #主成分の絞り込み pcaP <- pca pcaP$x[,-c(1:m)] <- 0 #相関行列PCAの逆計算 pca01 <- t(t(pcaP$x %*% t(pcaP$rotation)) * pcaP$scale + pcaP$center) #図示 par(mfcol = c(length(m1), length(m2)), family="HiraKakuProN-W3", omi=c(0.1, 0.1, 0.5, 0.1), mai = c(0.1, 0.1, 0.1, 0.1)) for(n in 1:p0){ a <- EBImage::Image(array(pca01[n,], dim=c(k, k, 3)), colormode = "Color") EBImage::display(a) if(n == 1){ mtext(side = 3, line=1, outer=T, text = paste0("PC", m, "までを用いる。"), cex=1.25) }} }}, movie.name = paste0("Img13_PCA.gif"), interval = 0.5, dpi=100, nmax = length(Num), ani.width = 500, ani.height=500, ani.type="png")
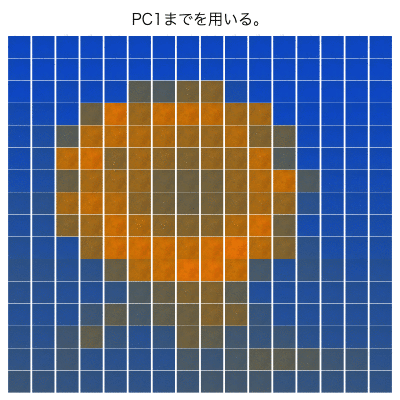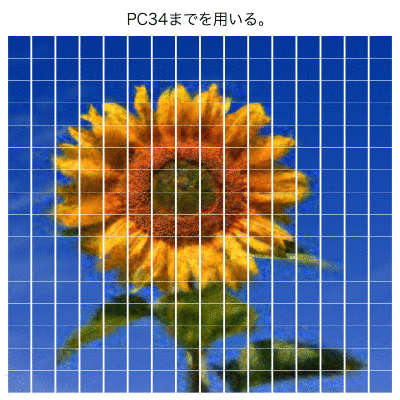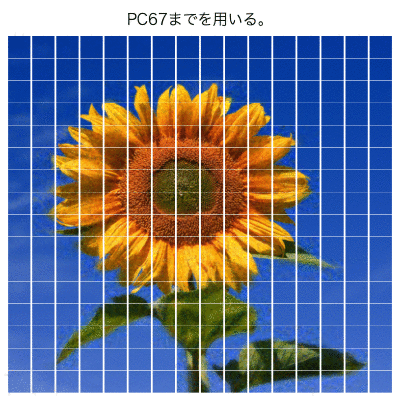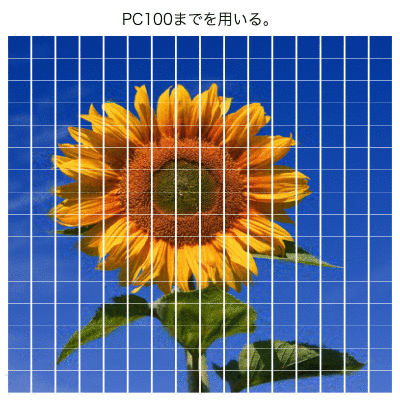
PC50くらいまでを使ったら、 ヒマワリ部分はほぼ再現できているように見える。
実行・生成に、結構時間がかかる。。
まとめ
画像データのPCA逆計算による画像圧縮を扱った。
PCAは様々なデータで応用できるのでホント奥が深い。
補足
分散共分散行列PCAの場合の逆計算
#Q-mode PCA: 分散共分散行列 pcaF <- prcomp(ImgDatCrop, scale = FALSE) #分散共分散行列PCAの逆計算 t(t(pcaF$x %*% t(pcaF$rotation)) + pcaF$center)
既に作成したPCAモデルを用いた次元圧縮
ここでは、新たなデータセットに対して、既に作成したPCAモデルを用いて次元圧縮を行う実施例を紹介する。
library(explor) data(USArrests) #PCA pca_test <- prcomp(USArrests[6:50,], scale. = TRUE) pca_test$supi <- predict(pca_test, USArrests[1:5,]) #可視化 explor(pca_test)
参考資料
メイン
http://racco.mikeneko.jp/Kougi/2016a/IPPR/2016a_ippr07_slide_ho.pdf
http://www.cfme.chiba-u.jp/~haneishi/class/2009/20091118.pdf
サブ
http://manabukano.brilliant-future.net/document/text-PCA.pdfmanabukano.brilliant-future.net
https://statistics.co.jp/reference/software_R/statR_9_principal.pdfstatistics.co.jp
https://data-science.gr.jp/implementation/ida_r_pca.htmldata-science.gr.jp
https://data-science.gr.jp/implementation/ida_r_pca.htmldata-science.gr.jp
*1:主成分分析には、応用分野によって様々な呼び名がある。信号処理の分野では、主成分分析のことを「KL変換」と呼ぶらしい。