この記事は、2021年12月15日現在のWikipedia 英語記事「Coronavirus」の一部を日本語翻訳したものである。
この情報は、Wikipediaの英語ページのみにあり、まだ日本語ページは存在していません。結構しっかりした内容で発信するのに有用だと思いましたので、このwikipedia記事の日本語訳を作成することにしました。
概要
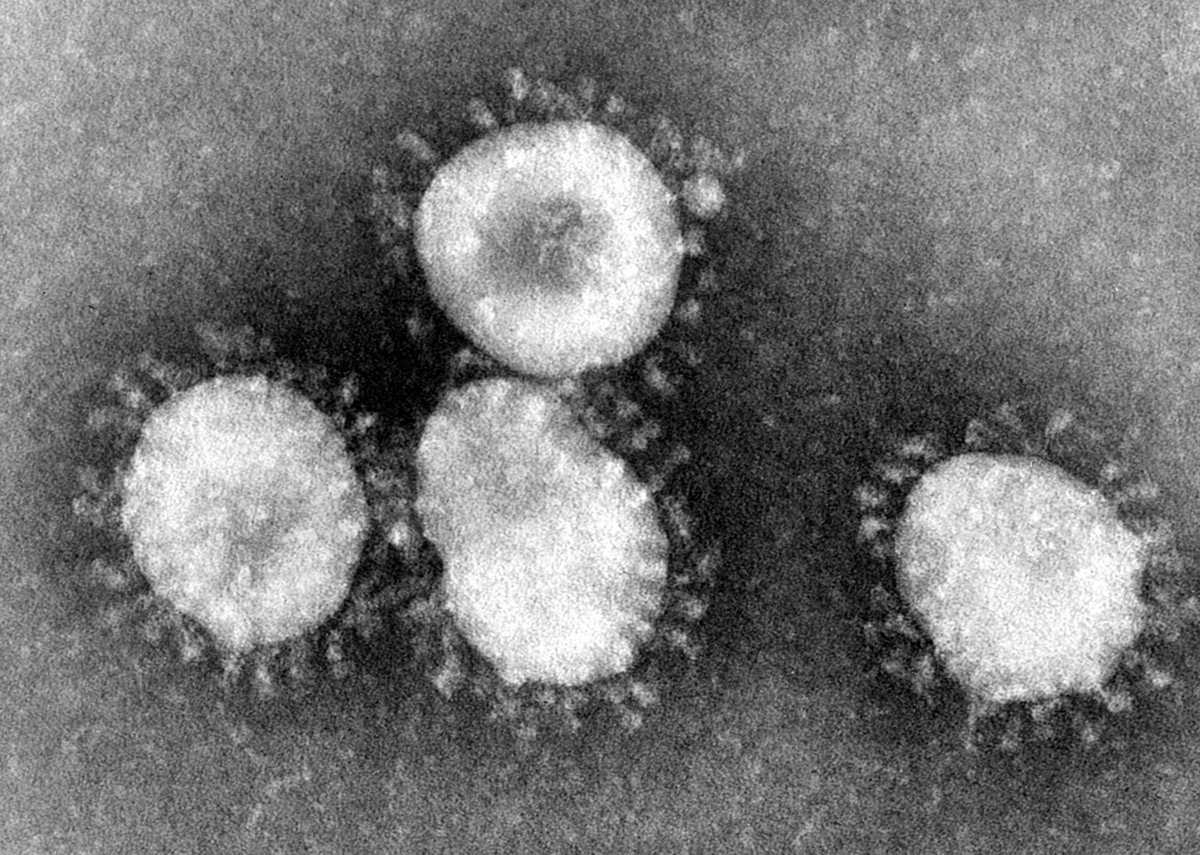
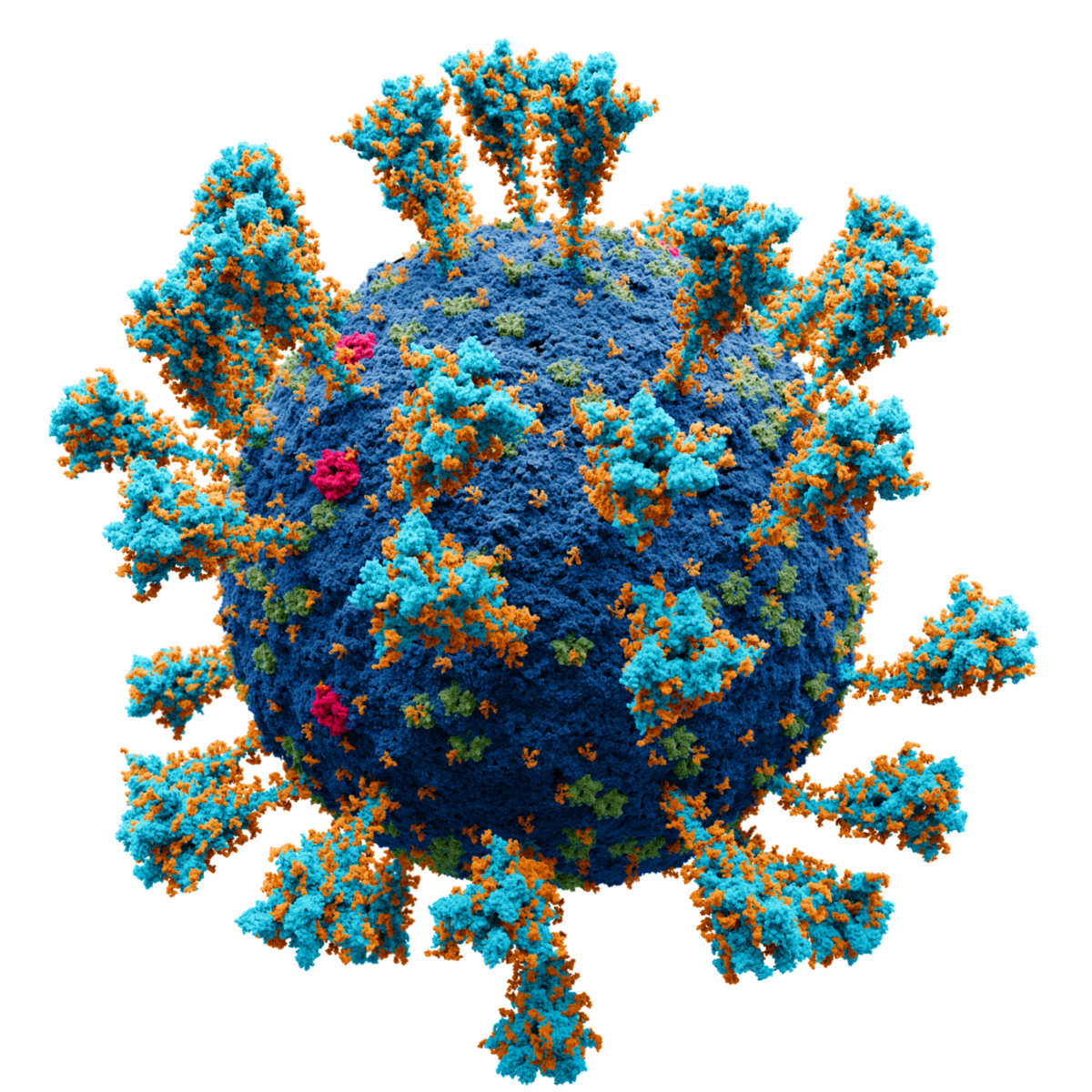
コロナウイルスは、哺乳類や鳥類に病気を引き起こす関連RNAウイルスの一群である。 ヒトや鳥類では、呼吸器感染症を引き起こし、軽度なものから致死的なものまで様々である。 ヒトの場合、風邪のような軽い症状から、SARS、MERS、COVID-19のような、より致死性のウイルスまで、さまざまな症状を引き起こす。牛や豚では下痢を起こし、マウスでは肝炎や脳脊髄炎を起こす。
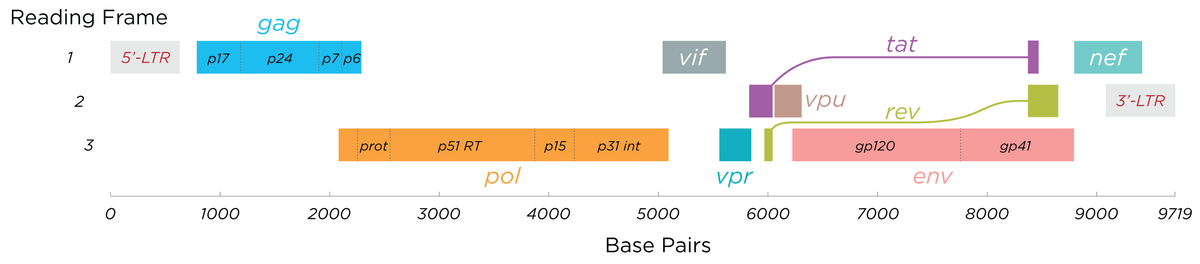
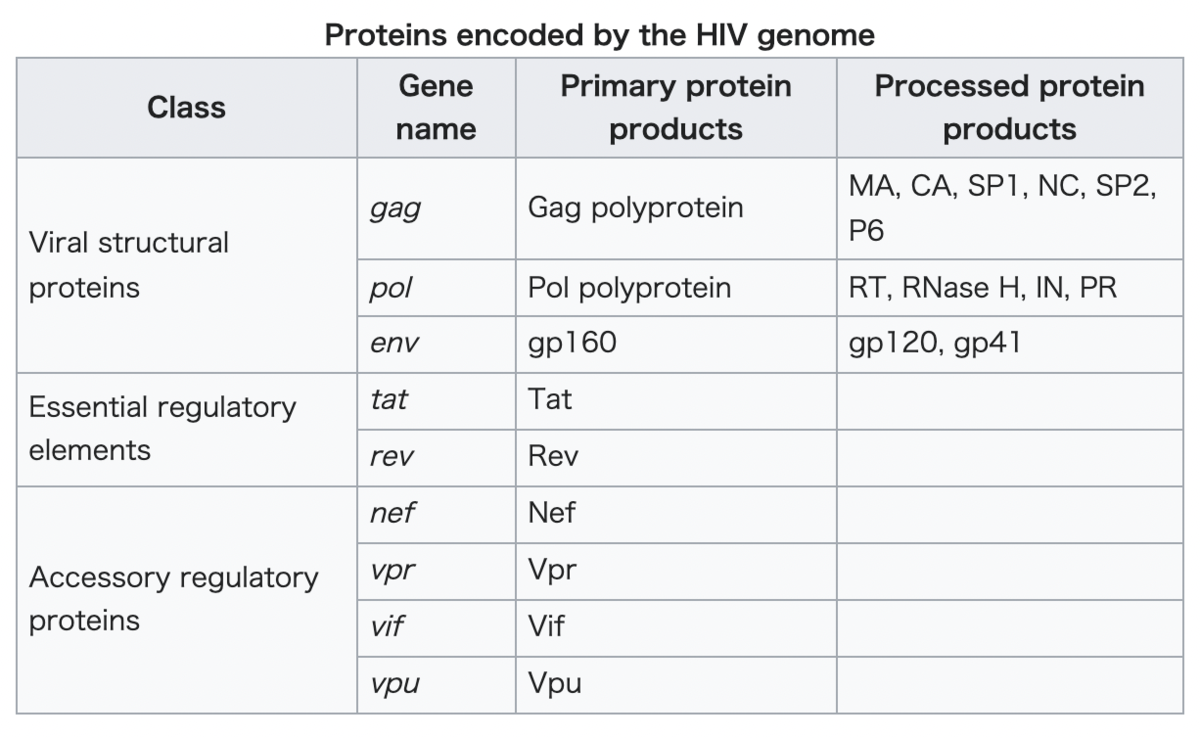
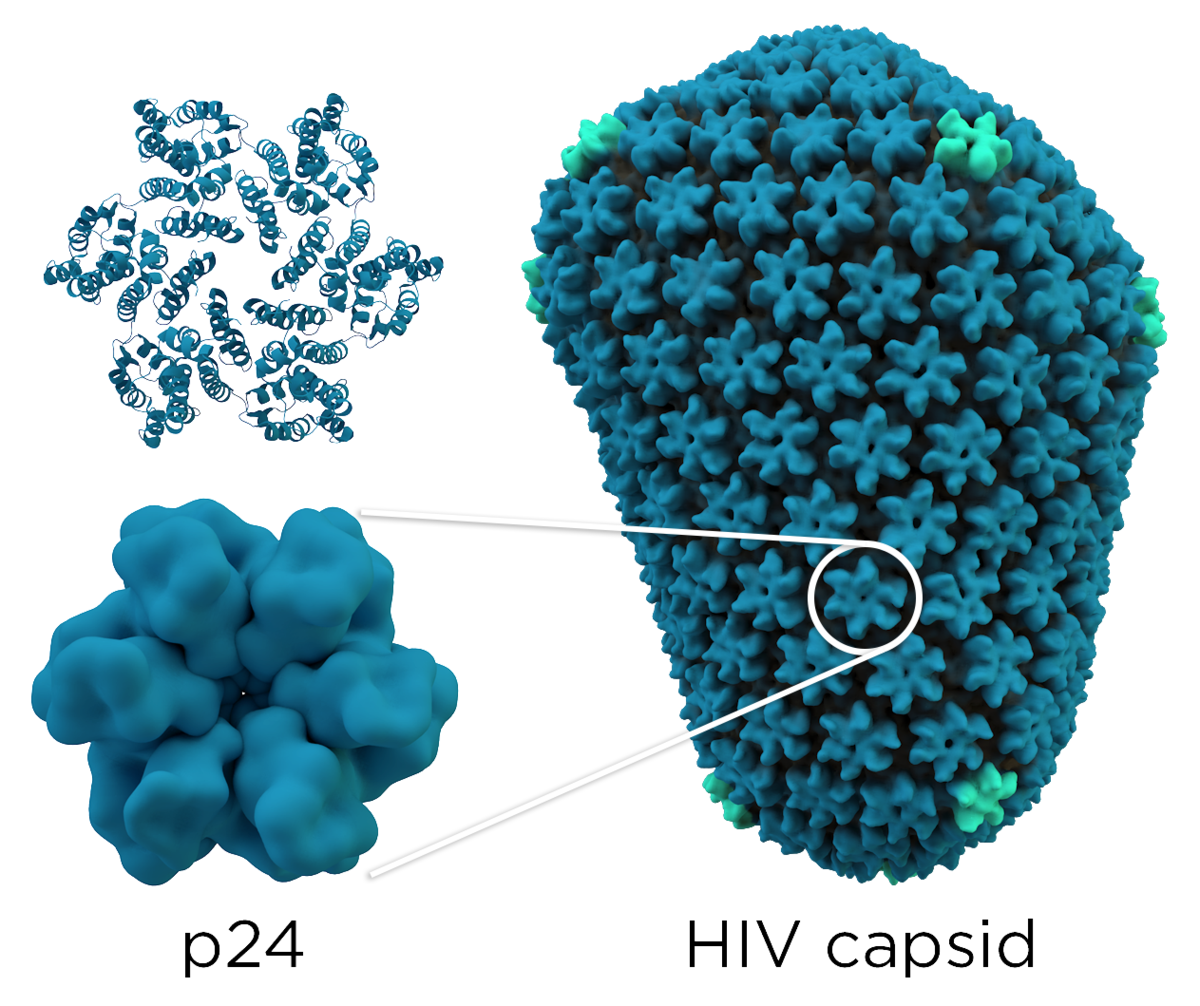
コロナウイルスは、コロナウイルス科、ニドウイルス目、リボウイルス科に属するオルトコロナウイルス亜科を構成する*1 *2。 ポジティブセンスの一本鎖RNAゲノムと、らせん対称のヌクレオキャプシドを持つエンベロープウイルスである*3。 コロナウイルスのゲノムサイズは、約26~32kb(キロベース)で、RNAウイルスの中では最大級である*4。 表面から棍棒状の突起が突き出ているのが特徴で、電子顕微鏡像では、名前の由来である太陽コロナを連想させるような像を示す*5。
Etymology / 語源
コロナウイルスという学名は、1971年に国際ウイルス命名委員会(後に、国際ウイルス分類委員会に改称)により属名として認められた*6。 新種の増加に伴い、2009年にアルファコロナウィルス、ベータコロナウィルス、デルタコロナウィルス、ガンマコロナウィルスの4属に分割された*7。 コロナウイルスという通称は、オルトコロナウイルス亜科のあらゆるメンバーを指すのに用いられる*8。 2020年現在、45種が公式に認識されている*9。
その2に続く
*1:"ICTV Taxonomy history: Orthocoronavirinae". International Committee on Taxonomy of Viruses (ICTV). Retrieved 2020-01-24.
*2:Fan Y, Zhao K, Shi ZL, Zhou P (March 2019). "Bat Coronaviruses in China". Viruses. 11 (3): 210. doi:10.3390/v11030210. PMC 6466186. PMID 30832341.
*3:Cherry J, Demmler-Harrison GJ, Kaplan SL, Steinbach WJ, Hotez PJ (2017). Feigin and Cherry's Textbook of Pediatric Infectious Diseases. Elsevier Health Sciences. p. PT6615. ISBN 978-0-323-39281-5.
*4:Woo PC, Huang Y, Lau SK, Yuen KY (August 2010). "Coronavirus genomics and bioinformatics analysis". Viruses. 2 (8): 1804–20. doi:10.3390/v2081803. PMC 3185738. PMID 21994708. Coronaviruses possess the largest genomes [26.4 kb (ThCoV HKU12) to 31.7 kb (SW1)] among all known RNA viruses (Figure 1) [2,13,16].
*5:Almeida JD, Berry DM, Cunningham CH, Hamre D, Hofstad MS, Mallucci L, McIntosh K, Tyrrell DA (November 1968). "Virology: Coronaviruses". Nature. 220 (5168): 650. Bibcode:1968Natur.220..650.. doi:10.1038/220650b0. [T]here is also a characteristic "fringe" of projections 200 A long, which are rounded or petal shaped ... This appearance, recalling the solar corona, is shared by mouse hepatitis virus and several viruses recently recovered from man, namely strain B814, 229E and several others.
*6:Lalchhandama K (2020). "The chronicles of coronaviruses: the bronchitis, the hepatitis and the common cold". Science Vision. 20 (1): 43–53. doi:10.33493/scivis.20.01.04.
*7:Carstens EB (2010). "Ratification vote on taxonomic proposals to the International Committee on Taxonomy of Viruses (2009)". Archives of Virology. 155 (1): 133–46. doi:10.1007/s00705-009-0547-x. PMC 7086975. PMID 19960211.
*8:Fan Y, Zhao K, Shi ZL, Zhou P (March 2019). "Bat Coronaviruses in China". Viruses. 11 (3): 210. doi:10.3390/v11030210. PMC 6466186. PMID 30832341.
*9:"International Committee on Taxonomy of Viruses (ICTV)". talk.ictvonline.org. Retrieved 2020-09-14.