
はじめに
Rでのネットワーク図の作成では、igraph packageがよく使われる。
ただ、igraphによるネットワーク図の描写は、1万ノードを超えたあたりから、結構な時間がかかる。
そのため、10万ノードを超えるような、大規模なネットワーク図の描画には、ちょっとしたコツがある。
今回、そんな大規模なネットワークの作成方法を取り上げ、実際の実行時間を検証してみた。
結論的には、igraph::plot.igraphを使うよりも、graphics::plot.defaultで描画する方が、10倍以上早くなる。
graphics::plot.defaultによるネットワーク図の作成例
まずは、Barabasi-Albertモデルで、サンプルのグラフ構造を作成する。
#パッケージ準備 library(igraph) #サンプル・グラフの作成 #sample_pa: Barabasi-Albertモデルによるスケールフリー・グラフの生成関数 #BAモデルは、グラフを構築するためのシンプルな確率的アルゴリズムである。 pa <- igraph::sample_pa(n=10, power=1, m=1, directed=F) pa #IGRAPH fe31e44 U--- 10 9 -- Barabasi graph #+ attr: name (g/c), power (g/n), m (g/n), zero.appeal (g/n), #| algorithm (g/c) #+ edges from fe31e44: #[1] 1-- 2 2-- 3 2-- 4 2-- 5 1-- 6 3-- 7 1-- 8 5-- 9 7--10 #graphics::plot.defaultによる作図 system.time(graphics::plot.default(layout_with_fr(pa), pch=20, cex=2.5, axes = F, type = "p", xlab = NA, ylab = NA)) #quartz.save(file = paste0("./Graph_net_00.png"), type = "png", dpi = 150); dev.off()

うーん、この図だと、ネットワークのエッジがない。
そこで、線分を追加するsegments関数で、エッジの描画を行う。
関数として組むと、こんな感じである。
このNetwork_plot関数で、pa を描画してみる。
#パッケージの準備 & 簡単な実行例 library(data.table) library(magrittr) source("https://gist.githubusercontent.com/kumeS/2c8204b1c8a78a16d00ec2eaed5a7ca5/raw/e67a5760d7fcfc3a9c8f62f729e407b2722f5bf2/Network_plot.R") pa <- igraph::sample_pa(n=10, power=1, m=1, directed=F) Network_plot(pa, Cex=2.5) #quartz.save(file = paste0("./Graph_net_01.png"), type = "png", dpi = 150); dev.off()
見慣れた、ネットワーク図が得られる。
グラフのレイアウトは、layout_with_fr *1 を使用している。
グラフ・ノード数による描画時間の比較
それでは、次に、グラフのノード数を増やしていって、igraph::plot.igraphとgraphics::plot.defaultを使う場合とでの実行時間を比較してみる。
検証したスクリプトは、以下の通りである。
##################### #10 nodes ##################### #plot.igraphでの実行 pa <- sample_pa(n=10, power=1, m=1, directed=F) system.time(plot.igraph(pa, vertex.size=10, vertex.label=NA)) # ユーザ システム 経過 # 0.014 0.002 0.019 #quartz.save(file = paste0("./Graph_01.png"), type = "png", dpi = 150); dev.off() #Network_plotでの実行 system.time(Network_plot(pa, Cex=2.5)) # ユーザ システム 経過 # 0.016 0.002 0.018 #quartz.save(file = paste0("./Graph_02.png"), type = "png", dpi = 150); dev.off()
##################### #100 nodes ##################### #plot.igraphでの実行 pa <- sample_pa(n=100, power=1, m=1, directed=F) system.time(plot.igraph(pa, vertex.size=4, vertex.label=NA)) # ユーザ システム 経過 # 0.023 0.003 0.026 #quartz.save(file = paste0("./Graph_03.png"), type = "png", dpi = 150); dev.off() #Network_plotでの実行 system.time(Network_plot(pa, Cex=1.5)) # ユーザ システム 経過 # 0.029 0.003 0.031 #quartz.save(file = paste0("./Graph_04.png"), type = "png", dpi = 150); dev.off()
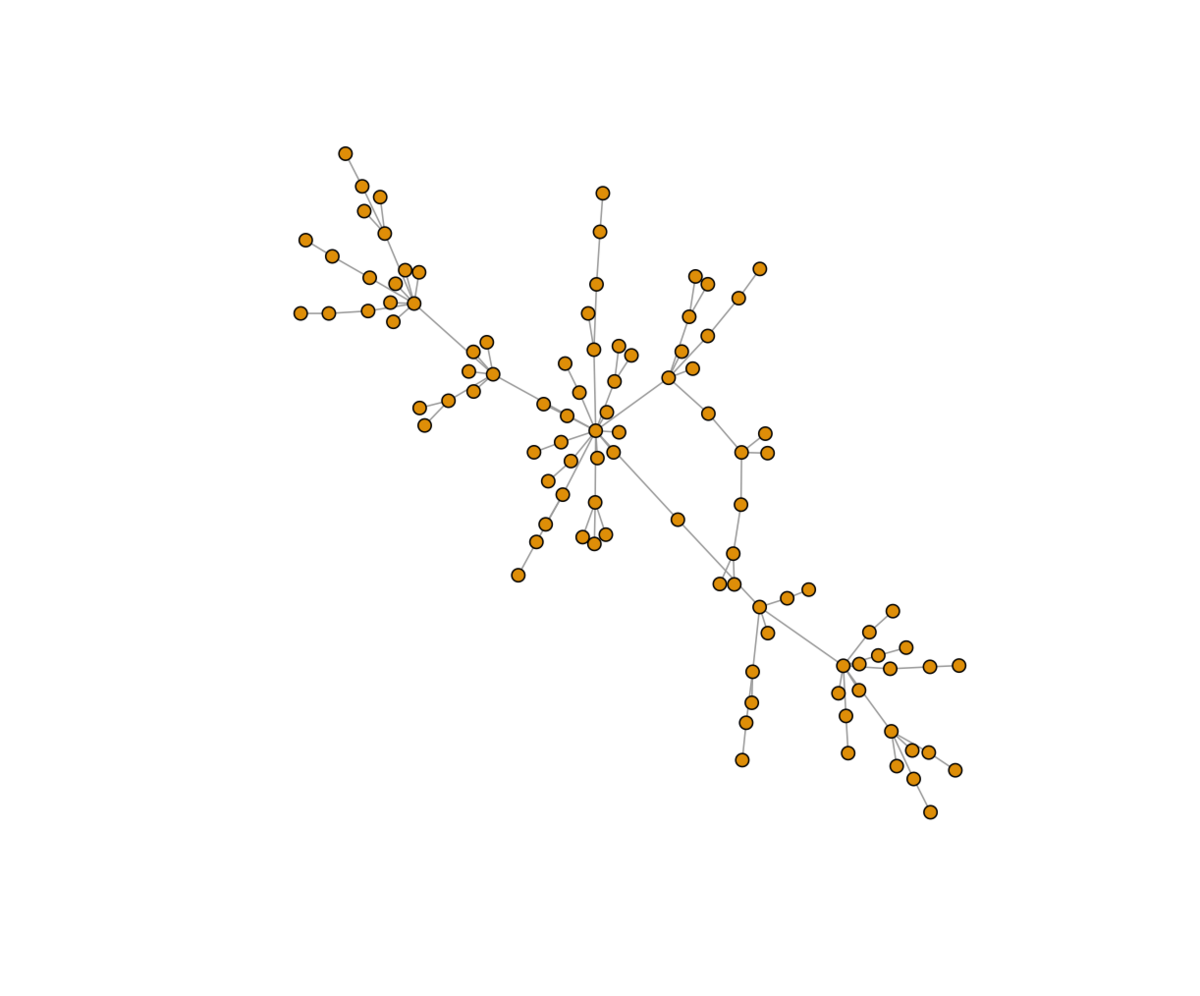
##################### #1000 nodes ##################### pa <- sample_pa(n=1000, power=1, m=1, directed=F) system.time(plot(pa, vertex.size=2, vertex.label=NA)) # ユーザ システム 経過 # 1.033 0.017 1.053 #quartz.save(file = paste0("./Graph_05.png"), type = "png", dpi = 150); dev.off() #Network_plotでの実行 system.time(Network_plot(pa, Cex=0.5)) # ユーザ システム 経過 # 0.771 0.006 0.781 #quartz.save(file = paste0("./Graph_06.png"), type = "png", dpi = 150); dev.off()


##################### #10000 nodes: 1万ノード ##################### pa <- sample_pa(n=10000, power=1, m=1, directed=F) system.time(plot(pa, vertex.size=1, vertex.label=NA)) # ユーザ システム 経過 # 11.014 0.035 11.061 #quartz.save(file = paste0("./Graph_07.png"), type = "png", dpi = 150); dev.off() #Network_plotでの実行 system.time(Network_plot(pa, Cex=0.2)) # ユーザ システム 経過 # 0.873 0.005 0.881 #quartz.save(file = paste0("./Graph_08.png"), type = "png", dpi = 150); dev.off()


##################### #100000 nodes: 10万ノード ##################### pa <- sample_pa(n=100000, power=1, m=1, directed=F) system.time(plot(pa, vertex.size=0.5, vertex.label=NA)) # ユーザ システム 経過 # 140.286 0.545 141.231 #quartz.save(file = paste0("./Graph_09.png"), type = "png", dpi = 150); dev.off() #Network_plotでの実行 system.time(Network_plot(pa, Cex=0.03)) # ユーザ システム 経過 # 9.381 0.021 9.423 #quartz.save(file = paste0("./Graph_10.png"), type = "png", dpi = 150); dev.off()

##################### #200000 nodes: 20万ノード ##################### pa <- sample_pa(n=200000, power=1, m=1, directed=F) system.time(plot(pa, vertex.size=0.5, vertex.label=NA)) # ユーザ システム 経過 # 278.272 0.539 279.129 #quartz.save(file = paste0("./Graph_11.png"), type = "png", dpi = 150); dev.off() #Network_plotでの実行 system.time(Network_plot(pa, Cex=0.01)) # ユーザ システム 経過 # 20.613 0.037 20.700 #quartz.save(file = paste0("./Graph_12.png"), type = "png", dpi = 150); dev.off()


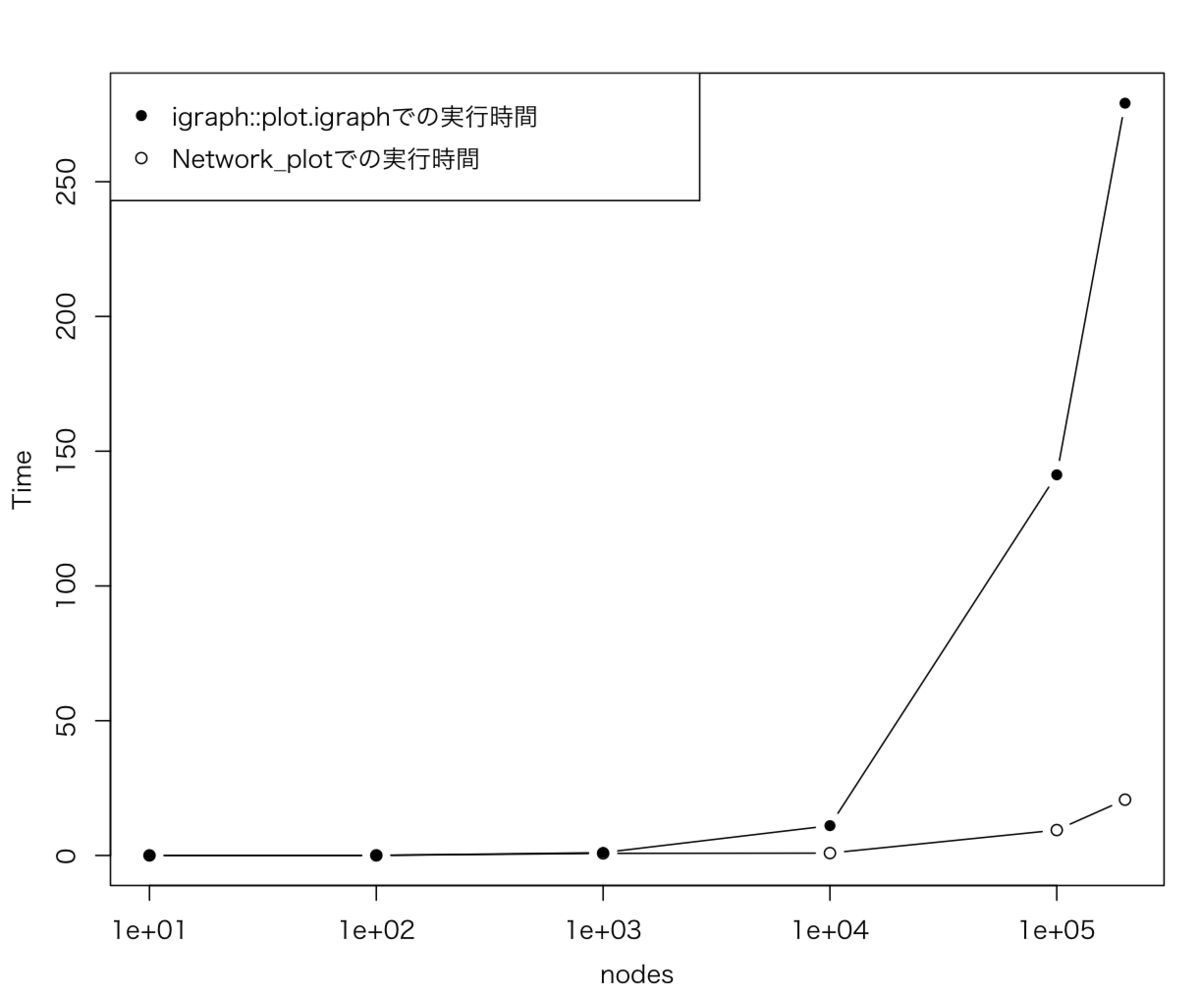
実行時間の可視化
実行時間を表で比較すると、1000や10000あたりから、すでに差が出てくる。
| ノード数 | igraph::plot.igraphでの実行時間 | Network_plotでの実行時間 |
|---|---|---|
| 10 | 0.019 | 0.018 |
| 100 | 0.026 | 0.031 |
| 1000 | 1.053 | 0.781 |
| 10000 | 11.061 | 0.881 |
| 100000 | 141.231 | 9.423 |
| 200000 | 279.129 | 20.700 |
図としてプロットすると、こんな感じ。
tm <- data.frame(a=c(10, 100, 1000, 10000, 100000, 200000), b=c(0.019,0.026,1.053,11.061,141.231,279.129), c=c(0.018,0.031,0.781,0.881,9.423,20.700)) par(family="HiraKakuProN-W3", mgp=c(2.5, 1, 0), mai=c(0.75, 0.75, 0.5, 0.25)) plot(tm[,1], tm[,2], type="n", log="x", xlab="nodes", ylab="Time") points(tm[,1], tm[,2], type="b", pch=16) points(tm[,1], tm[,3], type="b", pch=21) legend("topleft", legend=c("igraph::plot.igraphでの実行時間","Network_plotでの実行時間"), pch=c(16, 21), cex=1) #quartz.save(file = paste0("./Graph_13.png"), type = "png", dpi = 150); dev.off()
まとめ
描画速度が歴然と違う、、、igraphオブジェクトは1万ノードくらいを扱うのが良いところかな。
大規模なネットワークを扱いたい人には、graphics::plot.defaultが基本型だろう。
参考資料
*1:Fruchterman and ReingoldによるForce-Directed Layoutアルゴリズムを用いて、平面上に頂点を配置するレイアウト。