この記事は、2021年12月15日現在のWikipedia 英語記事「Coronavirus」の一部を日本語翻訳したものである。
この情報は、Wikipediaの英語ページのみにあり、まだ日本語ページは存在していません。結構しっかりした内容で発信するのに有用だと思いましたので、このwikipedia記事の日本語訳を作成することにしました。
Microbiology / 微生物学
Structure / 構造について

コロナウイルスは、独特の表面突起を持つ、大きなほぼ球状の粒子である*1。 その大きさは非常に多様で、平均直径は80〜120 nmである。 極端なサイズとしては、直径50~200 nmが知られている*2。 総分子量は、平均で40,000 kDaである。 コロナウイルスは、多数のタンパク質分子で埋め尽くされたエンベロープに包まれている*3。 脂質二重層のエンベロープ、膜タンパク質、ヌクレオキャプシドによって、ウイルスが宿主細胞の外にいるときは保護されている*4。
ウイルスエンベロープは、膜(M)、エンベロープ(E)、スパイク(S)の構造タンパク質が固定された脂質二重層で構成されている*5。 脂質二重層におけるE : S : Mのモル比は、約1:20:300である*6。 EとMタンパク質は、脂質二重層と結合して、ウイルスエンベロープを形成し、その大きさを維持するための構造タンパク質である*7。 Sタンパク質は、宿主細胞との相互作用に必要である。 しかし、ヒトコロナウイルスNL63は、Sタンパク質ではなく、 Mタンパク質が宿主細胞との結合部位を持つという特殊なタイプである*8。 エンベロープの直径は、85 nmである。 電子顕微鏡写真では、ウイルスのエンベロープは、明確な一対の電子密度の高い殻(ウイルス粒子の走査に用いられる電子ビームに対して比較的不透明な殻)として見える*9 *10。
Genome / ゲノム
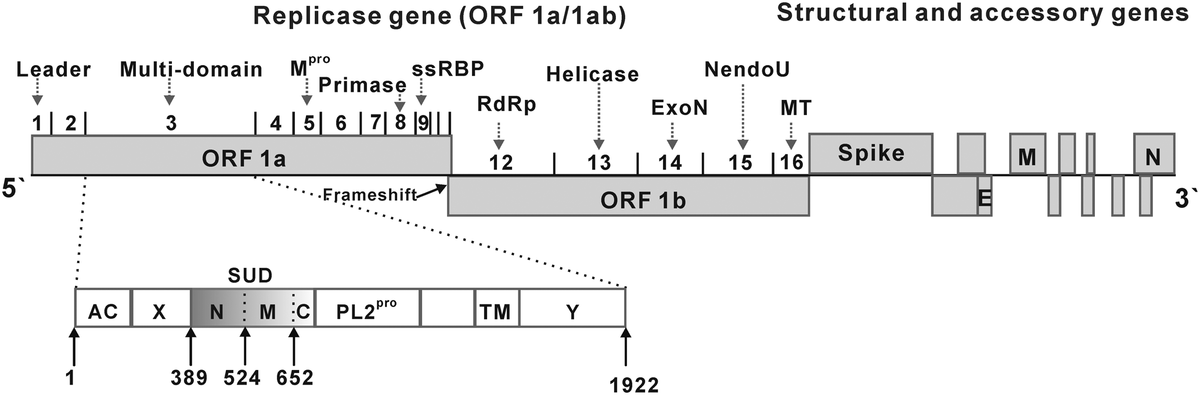
コロナウイルスは、ポジティブセンスの一本鎖RNAゲノムを持つ。 コロナウイルスのゲノムのサイズは、26.4から31.7kb(キロベース)である*11。 RNAウイルスの中では、コロナウイルスは最大級のゲノムサイズである。 ゲノムは、5' メチル化キャップと3' ポリアデニル [poly(A)] 化テールを持つ*12.
コロナウイルスのゲノム構成は、5′-leader-UTR-リプリカーゼ(ORF1ab) - スパイク(S) - エンベロープ(E) - 膜(M) - ヌクレオカプシド(N) - 3′UTR - poly (A) テールとなっている。 ゲノムの2/3を占めるオープンリーディングフレーム 1a と 1b は、レプリカーゼポリタンパク質(pp1ab)をコードしている。 レプリカーゼポリタンパク質は自己切断して、16個の非構造タンパク質(nsp1-nsp16)を形成する*13。
後半のリーディングフレームは、スパイク、エンベロープ、膜、ヌクレオキャプシドという4つの主要な構造タンパク質をコードしている*14。 これらのリーディングフレーム(ORF)の間に、 アクセサリータンパク質のORFが挟まれている。 アクセサリータンパク質の数とその機能は、 特定のコロナウイルス種に依存して独特である*15。
Replication cycle / 複製サイクル
Cell entry / 細胞への侵入
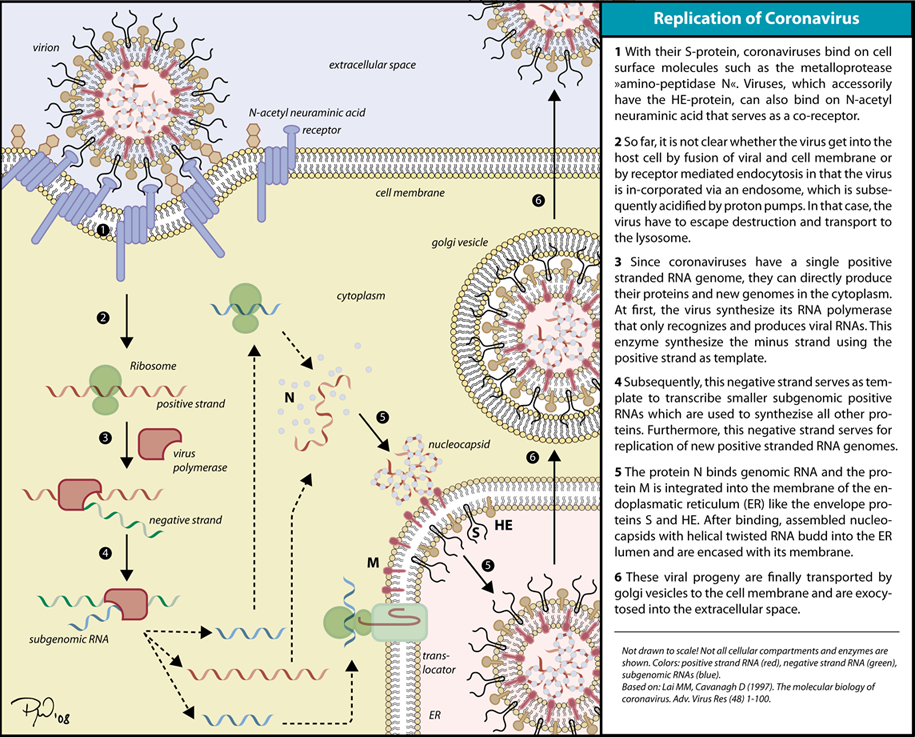
ウイルスのスパイクタンパク質が、宿主細胞の受容体と相補的に結合することで感染が始まる。 ウイルスが付着後、宿主細胞のプロテアーゼが受容体に付着したスパイクタンパク質を切断し、活性化させる。 宿主細胞のプロテアーゼが利用可能かどうかに応じて、切断と活性化により、ウイルスはエンドサイトーシスまたはウイルスエンベロープと宿主膜の直接融合によって宿主細胞内に侵入する*16。
*1:Goldsmith CS, Tatti KM, Ksiazek TG, Rollin PE, Comer JA, Lee WW, et al. (February 2004). "Ultrastructural characterization of SARS coronavirus". Emerging Infectious Diseases. 10 (2): 320–6. doi:10.3201/eid1002.030913. PMC 3322934. PMID 15030705. Virions acquired an envelope by budding into the cisternae and formed mostly spherical, sometimes pleomorphic, particles that averaged 78 nm in diameter (Figure 1A).
*2:Masters PS (2006). "The molecular biology of coronaviruses". Advances in Virus Research. 66: 193–292. doi:10.1016/S0065-3527(06)66005-3. ISBN 9780120398690. PMC 7112330. PMID 16877062.
*3:Lalchhandama K (2020). "The chronicles of coronaviruses: the electron microscope, the doughnut, and the spike". Science Vision. 20 (2): 78–92. doi:10.33493/scivis.20.02.03.
*4:Neuman BW, Kiss G, Kunding AH, Bhella D, Baksh MF, Connelly S, et al. (April 2011). "A structural analysis of M protein in coronavirus assembly and morphology". Journal of Structural Biology. 174 (1): 11–22. doi:10.1016/j.jsb.2010.11.021. PMC 4486061. PMID 21130884. See Figure 10.
*5:Lai MM, Cavanagh D (1997). "The molecular biology of coronaviruses". Advances in Virus Research. 48: 1–100. doi:10.1016/S0065-3527(08)60286-9. ISBN 9780120398485. PMC 7130985. PMID 9233431.
*6:Godet M, L'Haridon R, Vautherot JF, Laude H (1992). "TGEV corona virus ORF4 encodes a membrane protein that is incorporated into virions". Virology. 188 (2): 666–75. doi:10.1016/0042-6822(92)90521-p. PMC 7131960. PMID 1316677.
*7:Fehr AR, Perlman S (2015). "Coronaviruses: an overview of their replication and pathogenesis". In Maier HJ, Bickerton E, Britton P (eds.). Coronaviruses. Methods in Molecular Biology. 1282. Springer. pp. 1–23. doi:10.1007/978-1-4939-2438-7_1. ISBN 978-1-4939-2438-7. PMC 4369385. PMID 25720466. See section: Virion Structure.
*8:Naskalska A, Dabrowska A, Szczepanski A, Milewska A, Jasik KP, Pyrc K (October 2019). "Membrane Protein of Human Coronavirus NL63 Is Responsible for Interaction with the Adhesion Receptor". Journal of Virology. 93 (19). doi:10.1128/JVI.00355-19. PMC 6744225. PMID 31315999.
*9:Neuman BW, Adair BD, Yoshioka C, Quispe JD, Orca G, Kuhn P, et al. (August 2006). "Supramolecular architecture of severe acute respiratory syndrome coronavirus revealed by electron cryomicroscopy". Journal of Virology. 80 (16): 7918–28. doi:10.1128/JVI.00645-06. PMC 1563832. PMID 16873249. Particle diameters ranged from 50 to 150 nm, excluding the spikes, with mean particle diameters of 82 to 94 nm; Also See Figure 1 for double shell.
*10:Fehr AR, Perlman S (2015). "Coronaviruses: an overview of their replication and pathogenesis". In Maier HJ, Bickerton E, Britton P (eds.). Coronaviruses. Methods in Molecular Biology. 1282. Springer. pp. 1–23. doi:10.1007/978-1-4939-2438-7_1. ISBN 978-1-4939-2438-7. PMC 4369385. PMID 25720466. See section: Virion Structure.
*11:Woo PC, Huang Y, Lau SK, Yuen KY (August 2010). "Coronavirus genomics and bioinformatics analysis". Viruses. 2 (8): 1804–20. doi:10.3390/v2081803. PMC 3185738. PMID 21994708. Coronaviruses possess the largest genomes [26.4 kb (ThCoV HKU12) to 31.7 kb (SW1)] among all known RNA viruses (Figure 1) [2,13,16].
*12:Fehr AR, Perlman S (2015). "Coronaviruses: an overview of their replication and pathogenesis". In Maier HJ, Bickerton E, Britton P (eds.). Coronaviruses. Methods in Molecular Biology. 1282. Springer. pp. 1–23. doi:10.1007/978-1-4939-2438-7_1. ISBN 978-1-4939-2438-7. PMC 4369385. PMID 25720466. See section: Virion Structure.
*13:Fehr AR, Perlman S (2015). "Coronaviruses: an overview of their replication and pathogenesis". In Maier HJ, Bickerton E, Britton P (eds.). Coronaviruses. Methods in Molecular Biology. 1282. Springer. pp. 1–23. doi:10.1007/978-1-4939-2438-7_1. ISBN 978-1-4939-2438-7. PMC 4369385. PMID 25720466. See section: Virion Structure.
*14:Snijder EJ, Bredenbeek PJ, Dobbe JC, Thiel V, Ziebuhr J, Poon LL, et al. (August 2003). "Unique and conserved features of genome and proteome of SARS-coronavirus, an early split-off from the coronavirus group 2 lineage". Journal of Molecular Biology. 331 (5): 991–1004. doi:10.1016/S0022-2836(03)00865-9. PMC 7159028. PMID 12927536. See Figure 1.
*15:Fehr AR, Perlman S (2015). "Coronaviruses: an overview of their replication and pathogenesis". In Maier HJ, Bickerton E, Britton P (eds.). Coronaviruses. Methods in Molecular Biology. 1282. Springer. pp. 1–23. doi:10.1007/978-1-4939-2438-7_1. ISBN 978-1-4939-2438-7. PMC 4369385. PMID 25720466. See section: Virion Structure.
*16:Simmons G, Zmora P, Gierer S, Heurich A, Pöhlmann S (December 2013). "Proteolytic activation of the SARS-coronavirus spike protein: cutting enzymes at the cutting edge of antiviral research". Antiviral Research. 100 (3): 605–14. doi:10.1016/j.antiviral.2013.09.028. PMC 3889862. PMID 24121034. See Figure 2.